限制性内切核酸酶 — 分子剪
背景
术语“限制酶”源自Werner Arber和Matthew Meselson实验室对肠杆菌噬菌体λ(λ噬菌体)的研究。他们研究了某些大肠杆菌菌株通过酶促切割噬菌体DNA来抑制λ噬菌体活性的能力,而负责这种生长限制的酶就被称为限制酶。1, 2, 3
Werner Arber、Daniel Nathans和Hamilton O. Smith因其发现和表征了限制酶而于1978年获得了诺贝尔生理学或医学奖,自此开始了重组DNA技术的发展。
简介
限制酶也称为“分子剪”,因为它们在称为限制性位点的特定识别序列处或附近切割DNA。这些酶在两条DNA链的每一条上形成一个切口,因此亦被称为限制性内切核酸酶。4
病毒通过将DNA注入细胞来感染宿主细胞。这种病毒DNA劫持宿主细胞的病毒后代繁殖机制,导致宿主细胞死亡。为了克服病毒感染,许多细菌和古细菌已经进化出几种机制。主要的保护机制涉及使用限制酶通过在特定限制性位点切割来降解入侵的病毒DNA。与此同时,宿主细胞通过使用称为甲基化酶的其他酶来保护其自身的DNA不被切割,这种酶使得宿主识别序列内的腺嘌呤或胞嘧啶碱基甲基化。对于每种限制酶,宿主细胞产生相应的甲基化酶,其甲基化宿主DNA,并保护宿主DNA不被降解。这些酶构成限制-修饰(R-M)系统。
限制酶催化DNA的磷酸二酯骨架中3'-氧原子与磷原子之间的键的水解。酶需要Mg2+或其他二价离子保持其活性。
命名法
Smith和Nathans在1973年对限制性内切酶的命名规则进行了建议。根据这些原则,酶的名称以斜体三字母缩写词开头。第一个字母表示分离出酶的细菌属的第一个字母,第二和第三个字母则来自细菌种群。后面还可以跟随其他字母或数字来表示血清型或菌株。然后是一个空格和一个罗马数字,表示识别顺序。例如,Hind III表示是从流感嗜血杆菌(Haemophilus influenza)血清型d分离出的四种酶中的第三种。6
限制酶的类型
基于组分、切割位点的特征、以及辅因子要求,限制性内切核酸酶分为四类:I 型、II 型、III 型和 IV 型。
切割位点在表中以红色箭头进行表示。
影响限制酶活性的因素
取决于底物DNA和反应条件,限制酶显示出广泛的切割差异和可能的星号活性。为了获得所需的切割,对以下因素的控制变得非常重要:
- 星号活性:在非理想反应条件下,一些限制酶在不同于既定的识别序列的位点切割碱基序列。换句话说,它们在非特定位点切割。这种现象称为星号活性。诱导星号活性的一些因素包括:盐和甘油浓度过高、存在杂质、与底物DNA相比使用了过量的酶、孵育时间过长、或者缓冲液和辅因子不相容。
- 甲基化DNA:几个DNA分子在识别位点被甲基化,使它们对某些限制酶的切割具有抵抗性。例如,大多数大肠杆菌菌株表达Dam或Dcm甲基转移酶,其分别甲基化特定的识别位点而形成G6mATC和C5mCA/TGG。G6mATC对Mbo I的切割具有抵抗性。
- 温度:大多数内切酶进行靶标DNA消化的最适温度是37 °C。但是有一些例外,有些内切核酸酶的最佳温度比这更低或更高。例如,Taq I 在65℃下最佳消化,而Apa I(货号10899208001)在25℃下消化。
同裂酶和异裂酶6
同裂酶是具有相同识别序列和切割位点的限制酶。举例:Sph I (CGTAC/G)和Bbu I (CGTAC/G)

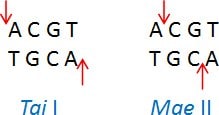
异裂酶是具有相同识别序列、但在该序列内部的不同位点切割DNA的限制酶。举例:Tai I (ACGT/)和Mae II (A/CGT)
限制性消化的产物
限制性消化双链DNA产生两种末端:粘性末端和平末端。
平末端具有促进连接的5'-磷酸基团。它们与其他平端DNA普遍相容。
由EcoR V产生的平末端

粘性末端是单链DNA短小链段,能够自我连接或与来自另一个DNA分子的互补区域连接。粘性末端具有含1-4个核苷酸的3'-或5'-悬突。
由Bln I(货号11558170001)产生的5’粘性末端

由Kpn I产生的3’粘性末端

缓冲液系列
我们的限制酶系列产品已经用五种独特的缓冲液进行了消化优化。当使用单一酶消化DNA时,请使用与酶一起提供的缓冲液。对于DNA的双重消化,则使用其中的两种酶均显示100%活性的缓冲液。或者,从常用双消化图表中确定最佳缓冲液。在某些情况下,由于缓冲液不相容(成分或温度),建议进行连续消化。关于用单一酶、两种限制酶以及连续DNA消化进行限制性消化的实验方案,请参见限制酶消化实验方案。选择正确的缓冲液对于获得两种酶的高活性和避免星号活性至关重要。我们提供五种缓冲液供用户选择相容的缓冲液进行所需的消化。大多数消化需要1-2种缓冲液,因此经济有效。
应用
限制性内切核酸酶在特定识别位点切割DNA的能力,使得这些酶具有广泛的应用,成为许多分子生物学技术的必要工具。下面是一些主要应用:
- 分子克隆:限制酶的一个流行应用是产生重组DNA分子。在这个过程中,限制酶切割供体DNA(通常是质粒)和载体DNA(通常是来自另一生物的基因),从而产生相容的末端。这些末端要么是“平的”,要么是“粘的”。两个被切割的DNA由称为DNA连接酶的酶连接在一起,产生重组DNA分子。然后,该重组DNA可被导入宿主生物体中进行复制。如需更多详细信息,请参阅限制酶克隆手册
- DNA作图,亦称为限制性作图,涉及使用限制性内切核酸酶来获得DNA片段或基因组的结构信息。作图过程涉及确定基因组中限制酶位点的顺序。拟将测定结构的目的DNA被用一系列限制性内切核酸酶切割,从而产生不同大小的DNA片段。这些片段在琼脂糖凝胶上被分离以后,即可确定目的DNA的结构。
基于特定DNA片段的已知限制酶位点,限制性内切核酸酶可用于验证该DNA片段的身份。 - 限制性标记基因组扫描是一种基因组分析方法,其利用限制酶的组合来显现给定生物体的整个基因组范围中甲基化水平的差异。这是识别任何DNA中偏离正常值的有用技术。它在检测肿瘤中高/低甲基化、基因的缺失或扩增、或整个生物体发育过程中基因表达的变化方面非常有效。8
- 基因测序:通过用限制酶消化大DNA分子,并用DNA测序仪处理所得到的片段,可以对大DNA分子进行测序。
- 限制性片段长度多态性(RFLP)涉及使用限制酶消化DNA样品,用凝胶电泳基于长度分离所得到的这些片段,并将它们转移到膜上。然后将这些片段与靶向被限制酶位点括起来的特定序列的放射性或荧光标记的探针结合。当所产生的片段长度各不相同时,即产生RFLP。每个个体都有一种称为“生物条形码”的独特图案。这项技术是第一个用于基因作图、遗传疾病基因定位、疾病风险测定、及亲子鉴定的DNA分析技术。9
- 脉冲场凝胶电泳涉及分离大的DNA片段,这些片段主要是用稀有切割限制酶消化细菌基因组产生的片段。所产生的独特图案用来区分不同的细菌菌株。对于比较普遍的疾病,可以用这项技术确定造成这种疾病的特定菌株。10, 11
- 基因表达系列分析(SAGE)是一种涉及以小标签形式定量和同时分析大量转录物的技术。限制酶在该技术中用作锚定酶和标记酶。12
- 限制酶介导整合(REMI)涉及使用限制酶在基因组中产生相容的粘性末端,用于插入已经用限制酶线性化的质粒DNA混合物中。质粒DNA与限制酶一起被转化到宿主细胞中,所述限制酶的作用是促进DNA整合到染色体中的同源限制性位点中。该技术可用于遗传筛选,以及在基因组中的特定点插入遗传和分子标记,以根据其突变表型鉴定目标基因。13
产品列表
参考文献
如要继续阅读,请登录或创建帐户。
暂无帐户?