SEQR – SeqPlex RNA扩增试剂盒实验方案
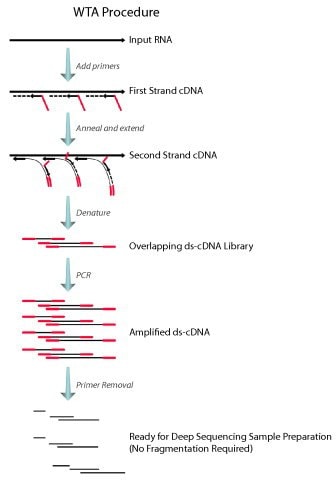
SeqPlex RNA扩增试剂盒可在进入常用深度测序平台工作流程之前为总RNA或分离后的mRNA提供一种扩增方法,在8小时或更短的时间内从低纳克至皮克级总RNA生成微克级的双链cDNA,适合对组织、培养细胞、福尔马林固定的样品或血清中分离的RNA(包括不含polyA尾的RNA)进行扩增,并同时保持未扩增样品中所发现的差异表达模式。
测序准备包括三个步骤。首先,用具有半简并3'末端以及指定通用5'末端的引物对样品RNA进行逆转录。随着DNA聚合进行,置换的单链可作为引物退火和延伸的新模板。接下来,由侧翼为单个通用引物序列的随机重叠片段组成的双链cDNA文库产物,在优化的PCR条件下扩增生成WTA(全转录组扩增)产物。从纳克级完整起始RNA开始,大部分的扩增产物大小范围为200至400 bp。对于受损的RNA和皮克级高质量RNA,可得到150至250 bp大小范围。最后,将SeqPlex扩增RNA与深度测序工作流程整合需要消除每个扩增子的引物序列。在样品制备之前需要完成有效的引物去除步骤。
除了深度测序外,SeqPlex扩增产品还可适用于芯片靶标或qPCR模板——无论是否进行引物去除
储存方法/稳定性
所有组分应储存在-20°C条件下。解冻使用时,所有组分应保存在冰上。
RNA处理技巧
该试剂盒中的试剂已经过测试确保不存在RNase。但用户必须穿戴基本的防护装备——包括手套和实验服——以保护实验结果的完整性。在整个过程中所有试剂的转移都应在层流罩或专用洁净室中进行。 冷冻RNA样品应在冰上进行解冻。
小心:
Sigma-Aldrich SeqPlex RNA扩增试剂盒 (SEQR)、SeqPlex DNA扩增试剂盒(SEQX)TransPlex® WTA试剂盒、完整全转录组扩增试剂盒(WTA2) 以及GenomePlex® WGA试剂盒中的某些成分具有相似命名。尽管它们功能大体相同,但不能替换使用。另外,熟悉WTA2试剂盒的用户请注意:
- SeqPlex RNA扩增试剂盒中的扩增预混液是5X溶液。
- PCR扩增设定包括最终的30分钟70 °C孵育。
针对下游测序的考虑因素
SeqPlex RNA扩增试剂盒可在扩增后高效去除引物序列。这些引物序列占扩增产物质量30-50%并可通过纯化除去。因此,引物去除反应的扩增产物加入量应是2倍测序所需量。
测序所需的核酸量具有平台特异性,Roche 454最低需要~1 µg起始核酸。Illumina MiSeq和HiSeq平台仅需50 ng,与ABI SoLiD一样。因此,此处提供的实验过程是基于最终引物去除反应后的1 µg产量。
为了确保可生成足够量的扩增RNA,请咨询您的测序服务供应商并对您的引物去除反应进行相应的放大。
操作流程
以100pg至5ng高质量总RNA(RIN > 8.0)开始时,单次扩增反应将产生2-4 mg扩增的双链cDNA。提高RNA模板起始量和质量通常会使产量增加。对于受损RNA——如从FFPE(福尔马林固定石蜡包埋)样品中分离的RNA——建议使用1-50 ng的起始RNA。 可以按比例放大或缩小反应体积以适应最终产物所需量的制备。
在扩增之前必须进行基因组DNA去除。如果这一步没有在RNA提取时完成,请按照操作说明以及最方便的文库合成规模使用产品AMPD-1 RNase-free DNase。
尽管扩增效率低于信使RNA,SeqPlex RNA 扩增试剂盒也可扩增核糖体RNA。尽管扩增效率低于信使RNA,SeqPlex RNA 扩增试剂盒也可扩增核糖体RNA。
5µL最终推荐反应体积的文库合成如下所描述。但通过对于处在早期指数扩增、扩增产量或β-actin转录水平(参见下面的“质量控制,产品保留”)的Ct值检测,2.5至25 µL的体积变化范围(同时保持75 µL扩增反应体积)并未显示出对于扩增的影响。确保对要比较的所有样品使用相同的文库合成体积。已提供足够的试剂用于试剂盒25 µL文库合成体积的指定数量反应。在推荐实验过程之后也提供了针对更高反应规模的可选设置建议。
文库合成
- 将文库合成缓冲液、文库合成溶液以及水进行解冻。颠倒或短暂涡旋混匀。如果文库合成溶液(L8670)存在沉淀,短暂加热至37 °C使其溶解。置于冰上。
- 按照单次反应的规格,将100 pg至5 ng的高质量完整总RNA或1-50 ng受损RNA(如FFPE或激光捕获样品RNA)与文库合成溶液相结合:
0.5 µL文库合成溶液(L8670)
加入无核酸酶水 (W4502) 至总体积3.3 uL。
- 吹打混匀并在热循环仪中将反应混合物在70℃下孵育5分钟,然后保持18 ℃。 不要在18 °C下保持10分钟以上。从热循环仪中取出反应物,放置在室温下或在接下来的步骤中保持18°C,但不要放在冰上。
注意:为了避免RNA复性以及可能的降解,请在18 °C或室温尽快完成第4、5步以及第6步的体系构建。
- 请在前述孵育步骤的同时制备以下预混液,并 立即将1.7 µL以下预混液与文库合成溶液中的变性RNA(步骤3)结合:
0.5 µL文库合成缓冲液(L9418)
0.8 µL水
0.4 µL文库合成酶(L9543)
- 吹打混匀并离心收集管壁上的溶液。
- 按照以下条件在热循环仪上进行孵育:
18 °C,10分钟
25 °C,10分钟
37 °C,30分钟
42 °C,10分钟
70 ºC,20分钟
4 °C,维持
- 通过离心进行冷凝液体收集。样品可立即用于扩增或储存在-20 ºC下,最长一个月。
扩增
- 5X扩增预混液将5X扩增预混液进行解冻。在混合后通过加热至37º C将所有可能存在的沉淀溶解。置于冰上。
- 按照单次反应的规格,将以下试剂转移至文库合成反应体系中*:
53.50 µL 无核酸酶水
15.00 µL 5X扩增预混液(A5112)
0.75 µL 1:1000的SYBR Green* 10 nM Tris-HCl溶液,pH 8.0(货号T3038)
) 0.75 µL扩增酶(A5237)
75 µL总反应体积
*可选择加入并未包含在试剂盒中SYGR Green(货号S9430),但在监测扩增时强烈推荐使用。 制备稀释液后立即加入混合液中。 在每次实验结束后丢弃稀释液。
- 吹打或短暂涡旋充分混匀。离心收集管盖和管壁上的残留溶液。
- 按照以下热循环程序进行PCR反应(强烈推荐使用实时qPCR):
1个循环
94° C,2分钟
17-19个循环*
94 °C,30秒
70 °C,5分钟(读取)
1个循环
70 °C,30分钟
注意:最终70 °C孵育30分钟对于引物去除以及下游测序应用至关重要。
*最佳扩增循环数随RNA起始量和质量而变化。正如通过实时定量PCR所观察到的,最佳的扩增一般都是进入扩增“平台期”后的2-3个循环内实现。通常,1 ng至5 ng的高质量RNA或10至50 ng的FFPE RNA需要~19个循环。在这种情况下,如果对扩增没有进行实时监测,则进行多次反应,否则无法实际监测单个扩增反应,而将扩增循环数设置为20。质量或量更低的RNA可能需要更高的起始量以及更多的扩增循环。如果扩增少于1 ng的RNA(RIN > 8.0)或少于10 ng的受损RNA(RIN < 8.0)且并没有实时监测,则进行多次反应,否则无法实际监测单个扩增反应,请设置扩增循环数为25。为了获得最佳的结果,请采用Sybr Green对扩增进行监测。
- 在完成热循环后,在4°C或-20°C维持反应体系等待至纯化。
- 为了去除残留的引物和核苷酸,请采用GenElute PCR Cleanup Kit(货号NA1020)根据试剂盒操作说明进行纯化。用50 µL无核酸酶水(而非试剂盒中的洗脱液)进行洗脱。如需要,可通过真空抽滤将洗脱液浓缩,但需要避免加热且不要让样品变得干燥。(因为扩增产物只是溶于水而并不含反离子,在干燥后样品会发生变性。变性会抑制引物去除的过程。)GenElute纯化柱的载量是10 ug,等同于常见的扩增反应纯化。
- 纯化的DNA可通过测量吸光度进行定量。每A260单位等同于50 ng/µL DNA。
引物去除
引物去除会造成少量或不会造成扩增产物损失。然而,建议采用加入了2 µg扩增产物的75 µL引物去除反应物以生成1µg的最终 产物,以进入深度测序的工作流程。(参见上文“针对下游测序的考虑因素”。)这一步可能会造成因为引物去除损失以及反应纯化过程中的其他任何额外损失。此外,还请对每个扩增的样品进行“无酶”对照反应。 需要对照反应来检测引物序列的去除(参见下文质检部分)。
注意:注:在进行引物去除后扩增产物不能再被进一步扩增。
注意:试剂盒中已含有用于75 uL“无引物去除酶”对照反应的足量试剂。 然而,您也可能会希望如下所示通过降低“无酶对照”体系规格来节省试剂及扩增产物。
注意:第15步中所描述的反应体系可用于引物去除反应以及(第16及17步中的)无酶对照。
- 将以下试剂混匀:
8.50 µL - 10x引物去除缓冲液(SR401)
1.70 µL -引物去除溶液(SR400)
X µL – 2.27 µg纯化后SeqPlex扩增产物(第14步)
Y µL - 水(W4502)
80.75 µL总反应体积
- 转移9.5 µL第15步中的混合液至另一不同的反应管中并加入0.5 µL的水(W4502)。这便是“无酶”反应物。吹打充分混匀。短暂离心以收集管盖和管壁上的残留溶液。
- 向剩余的71.25 uL溶液中加入3.75 uL引物去除酶(SR402)。这便是引物去除反应。吹打充分混匀。短暂离心以收集管盖和管壁上的残留溶液。
- 按以下程序进行引物去除以及无酶对照反应的孵育:
37 °C,60分钟
65 °C,20分钟
4 °C,保温
- 将样品从热循环仪中取出并短暂离心。
- 每种引物去除反应物及所有的无酶对照反应物各保留2 uL,用于下面的质检检测。置于冰上或储存在-20°C下,最长一个月。
- 按之前第13步所述,采用GenElute PCR Clean-up Kit将剩余引物去除反应产物进行纯化,或将纯化的样品储存在-20 °C下,最长一个月。
- 引物去除反应产物可通过测定吸光度进行定量。每A260单位等同于50 ng/µL DNA。
质量控制
扩增产物大小
对扩增后的完整总RNA进行Agilent Bioanalyis可显示出弥散以及范围在200至400 bp之间的主条带。受损RNA或低质量起始RNA的类似分析将会获得150至250 bp之间的范围。
引物去除
可采用5X扩增预混液和扩增酶结合qPCR预测引物去除效率。 可使用未纯化的引物去除反应以及相应的对照反应进行这些检测(第20步)。试剂盒中已含有用于引物去除反应以及“无引物去除酶”对照反应的15 uL qPCR检测所需的足量扩增预混液和酶。每种引物去除剂对照反应的1/1,000,000稀释液将用于该检测。引物去除的效率预计高于90%。
针对引物去除QC,将以下试剂的量用于单次qPCR反应:
3 µL 5X扩增预混液(A5112)
0.15 µL扩增酶(5237)
1.85 µL 1/10,000稀释液,SYBR的10 mM Tris-HCl溶液,pH 8.0(货号T3038)
10 µL 1/1,000,000 DNA稀释液(来自引物去除反应或无酶对照)
扩增条件:
1个循环
94 ° C,2.5分钟
40个循环
94 °C,30秒
70 °C,5分钟(读取)
理想的引物去除预计得到3-7的ΔCt结果:
(Ct)无引物去除酶 – (Ct)加上引物去除的酶
注意:可以在必要时进行单独的引物去除QC测试反应,以及制备试剂混合液的多个反应(推荐)。要移取指定少量溶液,请使用体积范围适当的移液器并使用硅化或低保留移液器吸头。在加样试剂前,确认通过离心将在储存过程中可能发生的任何冷凝液进行收集并通过涡旋、吹打或倒置正确混匀。
注意:已有结果表明通过该检测得到的预计引物去除比例与相应的测序结果之间存在关联。
扩增产物保留(可选)
为了证明在引物去除期间扩增产物保留,需要使用一对匹配相应源样品中已知表达的转录本的引物,进行额外的qPCR反应。在实验设置中需包括无酶对照反应(参见引物去除QC)。
(对于人类RNA样品,已知表达b-肌动蛋白时,可使用以下的引物)
5’-CGGGACCTGACTGACTACCTC-3’
5’-GAAGGAAGGCTGGAAGAGTGC-3’
- 不要设计引物对
- 引物对中的其中一条与下方立即显示的双链序列中上链的3’-端任意部分相匹配时
引物
3’ ←——————— 5’
5’-CTGAAGNNNNNNNNNNNNNNNN-3’
3’-GACTTCNNNNNNNNNNNNNN -5
- 或引物对扩增含有以下内部序列的双链DNA片段时
引物1
3’ ←————— 5’
5’-CTGAAGNNNNNNNNNNNNNNNN-3’
3’-GACTTCNNNNNNNNNNNNNN -5’
5’ —————→ 3’
引物2
- 引物对中的其中一条与下方立即显示的双链序列中上链的3’-端任意部分相匹配时
- 制备100 uM引物储存液。
- 制备由特定引物对以及2X SYBR Green Jumpstart Taq ReadyMix(货号S4438)所组成的混合液。
- 对于10次以上的反应:
- 确定2X SYBR Green Jumpstart Taq ReadyMix所需的总量(每次检测反应10 uL),记为x。
- 用x除以200,得到y。
- 两条引物各取y µL直接加入至x µL的2X SYBR Green Jumpstart Taq,通过吹打或涡旋充分混匀。
- 混合
10 µL 2X SYBR Green Jumpstart Taq ReadyMix +引物
10 µL引物去除反应或对照的1/1000稀释液
- 或者,对于10次以下反应:
- 用x除以20,得到z。
- 两条引物各取z µL加入,稀释至10 µM后直接加入至x µL的2X SYBR Green Jumpstart Taq,通过吹打或涡旋充分混匀。
- 按下述构建qPCR反应:
混合
11 µL 2X SYBR Green Jumpstart Taq ReadyMix +引物
9 µL引物去除产物或对照1/10000稀释液
- 对于10次以上的反应:
- 按照以下扩增条件进行qPCR反应:
1个循环
94 °C,2.5分钟
40个循环:
94 °C,30秒
__°C,30秒*
72 °C,30秒(读取)
溶解曲线,55 °C - 95 °C,每秒1度,每秒读取数据。
* TM具有引物特异性。
预计得到“+引物去除酶反应”的Ct值与“-引物去除酶反应”的Ct值相同,表明在进行引物去除后扩增反应产物完全保留。然而,通过光谱法检测则会因为引物去除而显示出30%至50%的质量损失。
深度测序
样品现在便可进入深度测序工作流程。SeqPlex扩增产物的主条带在200至400 bp范围内。尽管通常无必要,但也可通过超声DNA片段化仪器(如Covaris)或基于酶的片段化方法进一步片段化。
每条扩增子都具有5’-磷酸化以及3’端2个碱基的突出。在进行测序引物连接之前,采用T4聚合酶对双链片段的两端进行修复。用于Roche 454文库制备的连接产物已被报道包含有序列嵌合体。Illumina的3’-腺苷酸化可基本阻止这种情况的发生。当使用ABI SoLID测序前进行第二步的大小选择将有助于减少嵌合体发生概率。
扩增扩大化(可选)
之前所描述的5-uL“文库合成”是推荐的规格体积。对于不符合该规格的RNA样品体积,可使用以下的反应设置方法。
注意:有些情况下起始RNA量可能未知,例如RNA从极少量细胞中分离时。请注意到达平台期所需的循环数会随着起始RNA质量和数量提高(循环增加)或降低(循环减少)而变化。
下面的表格提供了第2步文库合成所需的最佳反应体积。
表2根据第4步文库合成按照表1中确定的反应扩大体积进行设置。
对于第9步扩增,将文库合成反应物全部加入至75-μL扩增反应物中,并相应调整水的体积(表3)。
产品列表
参考文献
如要继续阅读,请登录或创建帐户。
暂无帐户?