半胱氨酸蛋白酶——木瓜蛋白酶的特性和产品
E.C.3.4.22.2

分离自木瓜(番木瓜)乳
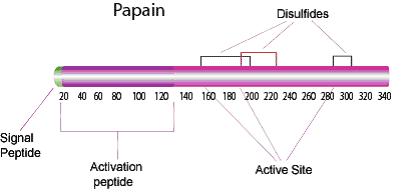
图 1.木瓜蛋白酶
特异性
木瓜蛋白酶可消化大部分蛋白底物,比胰蛋白酶范围更广。木瓜蛋白酶具有广泛的特异性,可断裂基础氨基酸,亮氨酸或甘氨酸的肽键。它还能水解脂类和酰胺类化合物。木瓜蛋白酶更倾向于P2位置有大的疏水侧链的氨基酸,它不接受P1'位置的缬氨酸。1
应用
- 木瓜蛋白酶广泛应用于细胞分离操作,在某些组织中已被证实比其他蛋白酶效率更高且破坏性更小。例如,木瓜蛋白酶可用于分离有活性的,形态完整的出生后大鼠的皮质神经元。2我们的木瓜蛋白酶(货号No. P4762)可用于分离平滑肌细胞3,4,能够分离出大量的有活性的平滑肌细胞,并且不影响细胞对刺激剂的敏感性5。
- 木瓜蛋白酶的部分水解作用可用于酶和其它蛋白质的结构研究。6-8
- 木瓜蛋白酶可应用于红细胞血清学,它通过对红细胞表面的修饰来加强或破坏许多红细胞抗原的反应性,可作为分类、抗体筛选、抗体鉴定的辅助手段。在血小板血清学中,木瓜蛋白酶也有应用。9
- 木瓜蛋白酶也可用于氨基酸、肽链和其它分子的酶合成。10-13
- Fab 和 F(ab')2抗体片段可用于不能出现Fc域的实验系统。在这种情况下,只用抗原结合片段(Fab)更为合适。木瓜蛋白酶可用于常规制备免疫球蛋白G的 Fab片段。免疫球蛋白M也可被木瓜蛋白酶消化,产生大量的同种Fab片段。15
- 木瓜蛋白酶将抗体切割为两个Fab片段(其可变区能够特异性识别抗原),和一个Fc片段。14它的切割位置在重链之间二硫键的铰链区之上,重链和轻链中间二硫键位点之下,从而形成了两个分开的Fab片段单价体(各包含单一抗体结合位点),和一个完整的Fc片段。各个片段可用凝胶过滤、离子交换或者亲和层析法来纯化。抗体消化和抗体片段提纯方法可参见《Antibodies: A Laboratory Manual》(E. Harlow and D. Lane, ed., Cold Spring Harbor Laboratory, Cold Spring Harbor, N.Y., 1988)。
- 在淋巴结、脾脏等组织,或是外周血制剂中,含Fc受体的细胞(如巨噬细胞、单核细胞、B淋巴细胞、自然杀伤细胞等)可结合完整抗体的Fc域,导致不含目标抗原的区域也出现背景染色。Fab片段的应用确保了抗体仅结合抗原而非Fc受体。这些片段也可用于血浆存在情况下的细胞染色,因为它们不能结合可溶解细胞的补体。Fab片段的应用使得目标抗原定位更精准,例如在电子显微镜的组织染色中。
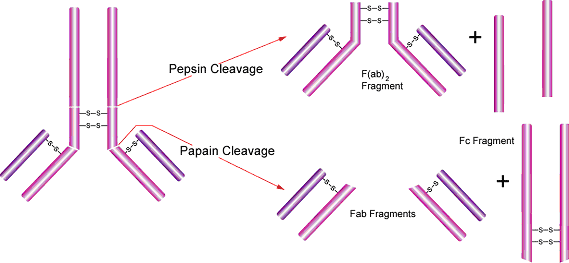
图 2.胃蛋白酶和木瓜蛋白酶切割
抑制剂
Loading
底物
Loading
溶解度和溶液稳定性
木瓜蛋白酶以10mg / ml溶于水中。使用之前,稀释于含5 mM L-半胱氨酸的缓冲液中。它的活化/稳定剂包括EDTA、半胱氨酸和二巯基丙醇。21
尽管木瓜蛋白酶溶液具有很好的温度稳定性,不过它的稳定性却依赖于pH。在酸性条件下,木瓜蛋白酶溶液并不稳定,例如在pH低于2.8时其活性大幅下降。在溶解状态下,木瓜蛋白酶的活性每天约丧失1-2%,可能是自我分解和/或氧化作用所致。
在分离过程中获得的木瓜蛋白酶常见的非活性形式,是蛋白质的活性位点巯基和游离半胱氨酸之间形成的混合二硫化物。23
木瓜蛋白酶溶液在几种变性剂中也保持稳定,例如经70%甲醇和8M尿素溶液再结晶之后依然保持完整活性。不过,经10%三氯乙酰或6M盐酸胍处理后木瓜蛋白酶的活性大幅下降。
相关产品
Loading
参考文献
1.
GP Moss. [Internet]. EC 3.4.22.2: International Union of Biochemistry and Molecular Biology (IUBMB).[updated 05 Jun 2020; cited 17 Jul 2020]. Available from: https://www.qmul.ac.uk/sbcs/iubmb/enzyme/EC3/4/22/2
2.
Kinoshita K, Sato K, Hori M, Ozaki H, Karaki H. 2003. Decrease in activity of smooth muscle L-type Ca2+ channels and its reversal by NF-?B inhibitors in Crohn's colitis model. American Journal of Physiology-Gastrointestinal and Liver Physiology. 285(3):G483-G493. https://doi.org/10.1152/ajpgi.00038.2003
3.
Driska SP, Laudadio RE, Wolfson MR, Shaffer TH. 1999. A method for isolating adult and neonatal airway smooth muscle cells and measuring shortening velocity. Journal of Applied Physiology. 86(1):427-435. https://doi.org/10.1152/jappl.1999.86.1.427
4.
HASEGAWA M, KOBAYASHI M, OYAMADA H, KAMISHIMA T, YOSHIDA C, OHATA H, MARUYAMA I, MOMOSE K, GOMI Y. 1987. Studies on isolated smooth muscle cells. IX Application of papain for isolation of single smooth muscle cells from guinea-pig taenia coli.. Jpn. J. Smooth Muscle Res.. 23(1):35-46. https://doi.org/10.1540/jsmr1965.23.35
5.
Margossian SS, Lowey S. 1973. Substructure of the myosin molecule. Journal of Molecular Biology. 74(3):301-311. https://doi.org/10.1016/0022-2836(73)90375-6
6.
Margossian SS, Lowey S. 1973. Substructure of the myosin molecule. Journal of Molecular Biology. 74(3):313-330. https://doi.org/10.1016/0022-2836(73)90376-8
7.
Shiozaki K, Yanagida M. 1991. A functional 125-kDa core polypeptide of fission yeast DNA topoisomerase II.. Mol. Cell. Biol.. 11(12):6093-6102. https://doi.org/10.1128/mcb.11.12.6093
8.
Kyer CI. 1995. Information technology law: What does the future hold?. Computer Law & Security Review. 11(3):140-142. https://doi.org/10.1016/s0267-3649(00)80035-7
9.
?-Nitro-?-amino acids as latent ?,?-dehydro-?-amino acid residues in solid-phase peptide synthesis. 2004(10):101. https://doi.org/10.3998/ark.5550190.0005.a11
10.
Rajesh M, Kapila S, Nam P, Forciniti D, Lorbert S, Schasteen C. 2003. Enzymatic Synthesis and Characterization ofl-Methionine and 2-Hydroxy-4-(methylthio)butanoic Acid (HMB) Co-oligomers. J. Agric. Food Chem.. 51(9):2461-2467. https://doi.org/10.1021/jf026093g
11.
Fukuoka T, Tachibana Y, Tonami H, Uyama H, Kobayashi S. 2002. Enzymatic Polymerization of Tyrosine Derivatives. Peroxidase- and Protease-Catalyzed Synthesis of Poly(tyrosine)s with Different Structures. Biomacromolecules. 3(4):768-774. https://doi.org/10.1021/bm020016c
12.
Burton SG, Cowan DA, Woodley JM. 2002. The search for the ideal biocatalyst. Nat Biotechnol. 20(1):37-45. https://doi.org/10.1038/nbt0102-37
13.
Greenfield E. 2014. Antibodies: A Laboratory Manual. 2. New York: Cold Spring Harbor Laboratory Press.
14.
NEWKIRK MM, EDMUNDSON A, WISTAR R, KLAPPER DG, CAPRA JD. 1987. A New Protocol to Digest Human IgM with Papain that Results in Homogeneous Fab Preparations that Can Be Routinely Crystallized. Hybridoma. 6(5):453-460. https://doi.org/10.1089/hyb.1987.6.453
15.
Mitchel R, Chaiken I, Smith E. 1970. The Complete Amino Acid Sequence of Papain. J. Biol. Chem. 2453485-3492.
16.
Smith EL, Kimmel JR, Brown DM. 1954. CRYSTALLINE PAPAIN: II. PHYSICAL STUDIES; THE MERCURY COMPLEX. J. Biol. Chem.. 207533-549.
17.
Sluyterman L, de Graaf M. 1972. The effect of salts upon the pH dependence of the activity of papain and succinyl-papain. Biochimica et Biophysica Acta (BBA) - Enzymology. 258(2):554-561. https://doi.org/10.1016/0005-2744(72)90247-1
18.
Glazer AN, Smith EL. 1961. Phenolic Hydroxyl Ionization in Papain. J. Biol. Chem.. 2362948-51.
19.
Pace CN, Vajdos F, Fee L, Grimsley G, Gray T. 1995. How to measure and predict the molar absorption coefficient of a protein. Protein Sci.. 4(11):2411-2423. https://doi.org/10.1002/pro.5560041120
20.
21.
Kilara A, Shahani KM, Wagner FW. 1977. Preparation and properties of immobilized papain and lipase. Biotechnol. Bioeng.. 19(11):1703-1714. https://doi.org/10.1002/bit.260191109
22.
Klein IB, Kirsch JF. 1969. The Activation of Papain and the Inhibition of the Active Enzyme by Carbonyl Reagents. J. Biol. Chem.. 2445928-5935.
登录以继续。
如要继续阅读,请登录或创建帐户。
暂无帐户?