MISSION® esiRNA规格
定制和预设计esiRNA
自2020年10月下旬/11月初开始,管装交付的esiRNA产品——单独的esiRNA、esiOPEN 和 esiSEC——将开始以冻干形式而非溶液形式发货(板装esiRNA 产品——esiLibrary 和 esiFLEX——将继续以溶液形式发货)。出于以下原因,我们正在对管装产品(大多数 esiRNA 管装发货)进行如下调整:
- 冻干材料可在环境温度下发货,而不是在干冰上冷冻。运输费用的减少将帮助我们将产品价格保持在尽可能低的水平。
- 发货包装中去除干冰,可让我们减少包装材料,进而帮助我们履行减少环境影响的承诺。作为 RNA,esiRNA 在室温下至少可稳定保存 3 周。收到您的 esiRNA 后,请在临用前 -20 °C 储存。产品技术公告中的附录 2 包含重悬方案。上述调整存在一段过渡期,部分 esiRNA 可能仍以溶液形式运输,部分会冻干发货。我们将尽快加速冻干发货完全转型。
所有订购和报价选项均包含在esiRNA规格表中
MISSION® esiRNA(Eupheria Biotech提供支持)为 RNAi 研究人员进行蛋白质编码基因和 lncRNA(长链非编码 RNA)的转录后沉默,提供了一种经过验证、实惠且简单的方法。生物制备的 esiRNA 由异源性 siRNA(天然 RNA,无修饰)混合物组成,靶向相同的 mRNA 序列。这些多重沉默触发器导致高度特异性、有效的基因敲低,脱靶效应低于单一的化学合成 siRNA(图 1)。

图 1.可通过化学合成的 siRNA 或酶法制备的 siRNA (esiRNA) 来敲低靶 mRNA。A) 化学合成的 siRNA 由一个与目标 mRNA 互补的 21 bp 沉默触发器组成。转染反应中高浓度的 siRNA 会导致明显的脱靶效应。B) 而esiRNA 由数百种 siRNA (21 bp) 的混合物组成,覆盖靶 mRNA 的 300 – 600 bp 区域。每中单独 siRNA 在混合物中的浓度较低,从而导致较低的脱靶效应,实现有效敲低。
产品优势
- 有保障的基因沉默效果
- 与化学合成的单一 siRNA 相比,脱靶效应更低
- 高靶向特异性造就高效的基础筛选工具(图 2)
- 实惠的基因组规模 RNAi 筛选工具
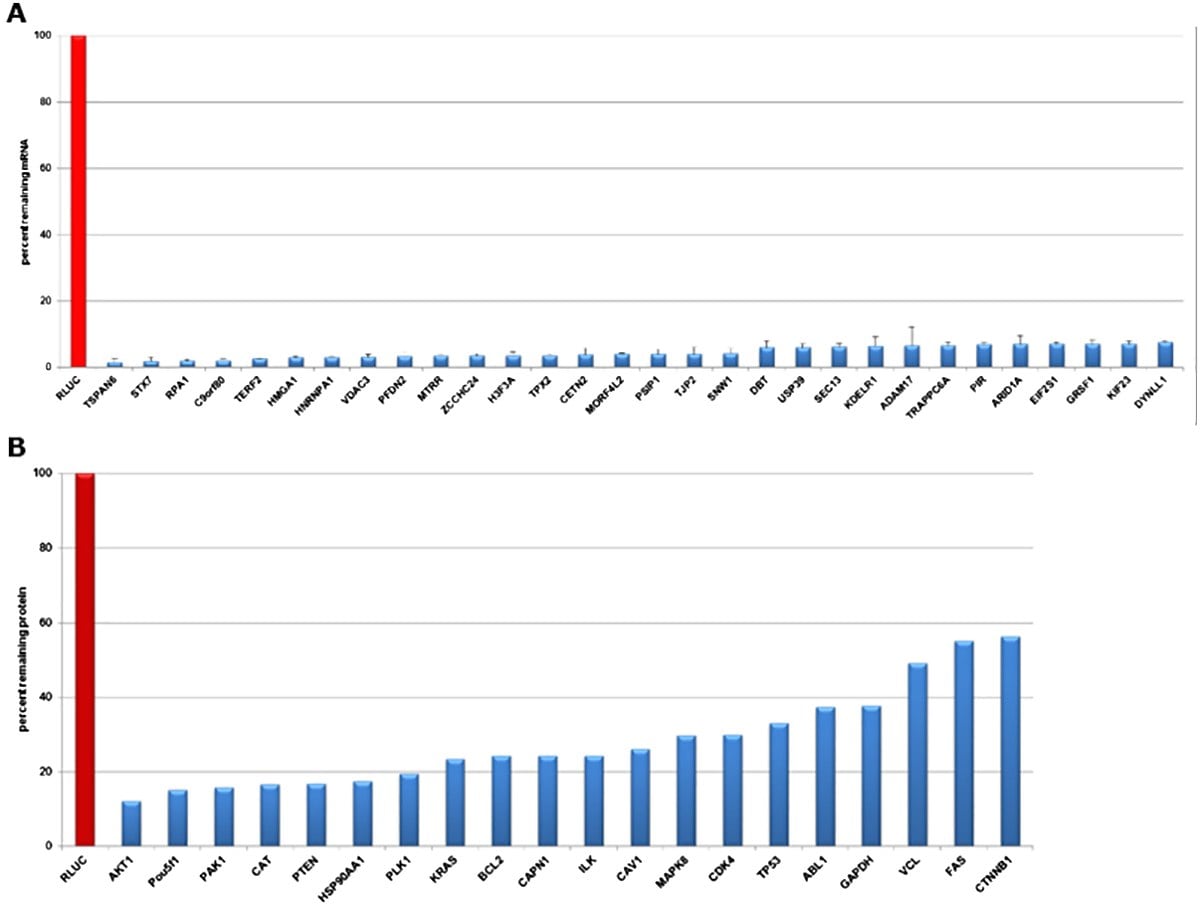
图 2.用靶向海肾萤光素酶(阴性对照)和不同表达靶标的 esiRNA 转染 HeLa 细胞,在 mRNA 和蛋白质水平评估基因敲低效果。A) 在转染后 24 小时通过qPCR 数据验证敲低效果。在所有转染细胞中,mRNA 丰度降低了 >90%。B) 在转染后 72 小时,通过定量蛋白质印迹数据验证有效敲低效果。蛋白水平降低了 45% 至 90%。
产品特性
以下说明适用于 esiRNA 规格表中包含的所有产品选项:
- 纯化方法:Q-Sepharose 分离、异丙醇沉淀和乙醇洗涤
- 序列形式:数百个平均双链长度为 21 bp 的 siRNA 混合物库
- 质检方法:分两个阶段进行
o 通过凝胶电泳和 DNA 测序分析 cDNA 克隆的 PCR 产物
o 通过凝胶电泳分析消化反应 - 稳定性:-20 °C 下可维持活力 2 年
如果下表中注明“咨询”,或者所列标准规格无法满足您的要求,请发送邮件至sirnarequest@milliporesigma.com进行咨询。
浏览完整的预设计esiRNA清单。
对照esiRNA
提供多种阳性和阴性对照(图 3):
- 阳性对照:人类Eg5 (KIF11)和小鼠Eg5 (Kif11) esiRNA 以及所有经过验证的 esiRNA(见下表),用于确保转染和优化实验构建
- 阴性对照:RLUC、FLUC和EGFP(EGFP 也可用作表达 EGFP 的细胞中的阳性对照)esiRNA,用于区分序列特异性沉默和非特异性效应
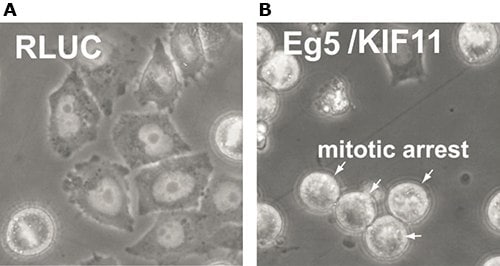
图 3.相对于 A) RLUC(阴性对照)和 B) Eg5/KIF11(阳性对照)的转染 esiRNA 的 HeLa 细胞表型分析。RLUC 不诱导任何表型变化,而 Eg5/KIF11 诱导有丝分裂停滞(显示圆形细胞形态)。
经验证的esiRNA
许多常见基因靶标已经过 ≥70% mRNA 敲低验证(qPCR和蛋白质免疫印迹验证数据)。请参阅下表,了解常订购的经验证esiRNA 清单(按基因符号排列)。经验证的esiRNA适于转染优化或用作阳性对照。
lncRNA esiRNA
转录组分析表明,高达 90% 的基因组被转录为非编码 RNA,其中包括 lncRNA。lncRNA 与不同的功能相关,包括染色质修饰、转录因子共激活、转录、与 RNA 结合蛋白的相互作用以及启动子抑制。此外,lncRNA 还与癌症等疾病有关。因此,我们有必要进一步阐明 lncRNA 的作用机制详情。esiRNA 是研究 lncRNA 功能的有效筛选工具(图 4)。
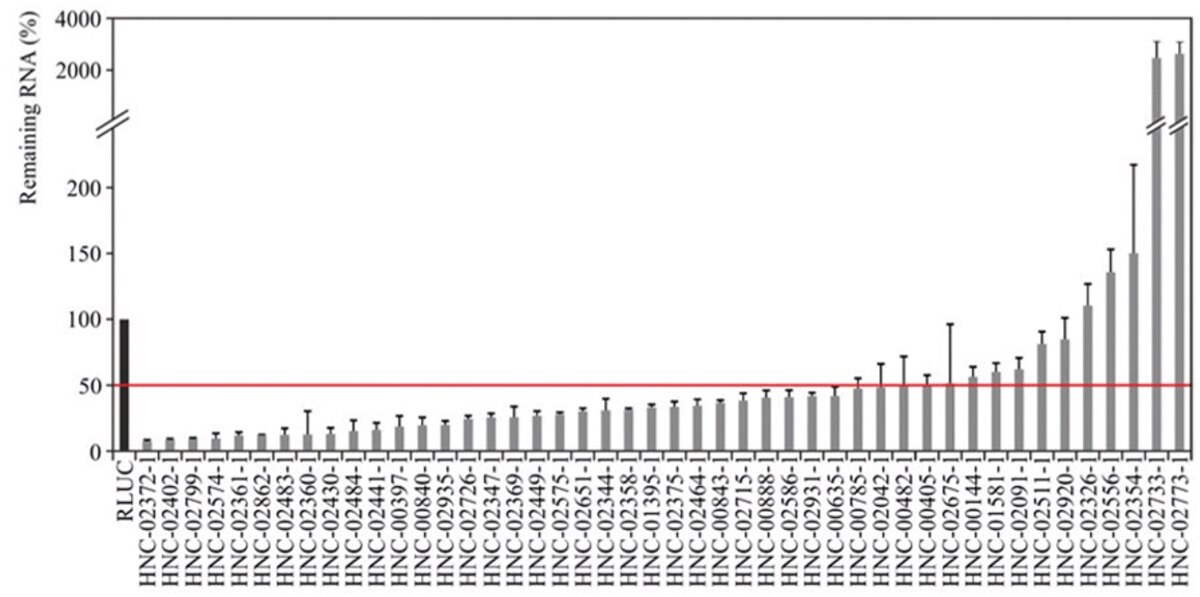
图 4.用相应 esiRNA 转染后 24 小时,通过 qPCR 数据验证 HeLa 细胞中的 lncRNA 敲低效果。用靶向 RLUC 的 esiRNA 转染对照并标准化结果。
产品制造
通过大肠杆菌RNase III内切核糖核酸酶切割长链dsRNA(双链 RNA),制得生物制备(图 5)的 esiRNA
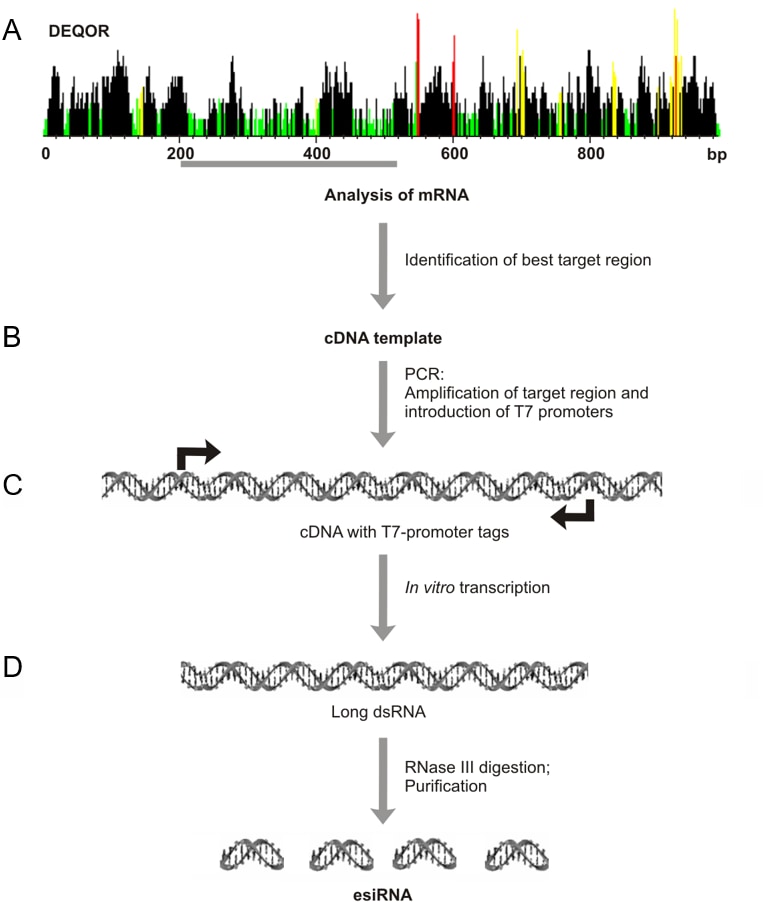
图 5.单基因 esiRNA 制造概览。A) 采用DEQOR,筛选产生最多数量高效 siRNA、覆盖所有已知转录本变异且脱靶效应最低的 mRNA 目标区域。B) 从 cDNA 克隆中提取目标区域,并通过 PCR 扩增(同时采用两种引物引入 T7 启动子。C) 使用 RNA 聚合酶体外转录PCR 产物。D) 用 RNase III 消解退火的长双链 RNA (dsRNA) ,纯化去除 DNA 模板、残留NTP 和未完全消解的dsRNA。最后,制得包含数百种siRNA、平均双链长度21 bp的esiRNA混合物。
esiRNA保证
转染浓度≥30 nM 时,MISSION® esiRNA可将培养细胞中的靶标mRNA水平降低70%。如果 esiRNA 未将靶标基因敲低70%,我们将针对该基因免费再提供一份esiRNA。如果我们未储备该基因的esiRNA,将全额退款。
上述保证需提供有关转染效率的适当支持数据。转染效率的适当支持数据包括 qPCR 数据,即将经验证的 MISSION® esiRNA 的靶标 mRNA 水平与适当的阴性对照(如模拟转染、RLUC、FLUC 或 eGFP esiRNA)进行对比,在 ≥ 30 nM 浓度转染的情况下经证靶标 mRNA 敲低率小于 70%。
由于抗体和蛋白质半衰期不定,我们不接受基于蛋白质的检测方法数据。
常问问题解答
有关 esiRNA 的更多信息,请访问我们的常见问题解答部分。
视频教程
了解有关使用 esiRNA 筛选哺乳动物细胞的信息。
如需其他帮助,请通过sirnarequest@sial.com咨询我们的技术服务团队。
了解更多有关RNA 干扰的信息:从单基因研究到全基因组筛查 - Julia Krüger,Eupheria Biotech公司高级科学家
精选引用文献
单基因esiRNA
全基因组和子库筛选
lncRNA esiRNA
如要继续阅读,请登录或创建帐户。
暂无帐户?