RNA免疫沉淀 Chip (RIP) 检测
蛋白质-RNA相互作用在细胞中具有重要作用,包括结构、催化和调节功能。RNA免疫沉淀(RIP)与染色质免疫沉淀(ChIP)相似,可用于检测单个蛋白质与特定核酸的结合,例如mRNA、非编码RNA(如长非编码RNA、增强子RNA、miRNA)和病毒RNA。RIP检测方法主要有两种:天然和交联。天然RIP检测可直接鉴定与目标蛋白质结合的RNA及其在免疫沉淀样品中的丰度。而交联RIP检测可精确定位蛋白质和RNA的直接和间接结合位点(图1)。在交联RIP分析中,使用高反应性甲醛处理活细胞,使得在体内非常接近的分子之间产生蛋白质-RNA可逆性交联(图2)。
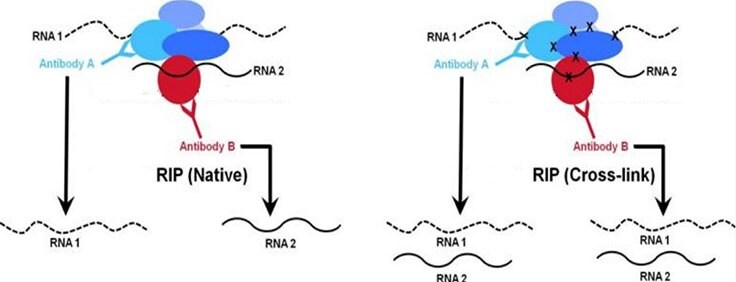
图 1.天然和交联RIP的比较尽管天然和交联方法都可以分析染色质相关RNA,但是天然RIP方法(左图)通常可以回收高亲和力的蛋白质-RNA相互作用。相反,交联方法(右图)经过设计可捕获更高分子量的复合物,并且更容易捕获较弱相互作用的RNA。
https://www.emdmillipore.com/US/en/product/EZ-Magna-RIP-RNA-Binding-Protein-Immunoprecipitation-Kit,MM_NF-17-701RNA 免疫沉淀 (RIP) 检测方案工作流程
您选择的RIP检测方案部分取决于蛋白质-DNA结合位置(细胞质或细胞核)。常规全细胞裂解物RIP方案可能最适用于细胞质相互作用,而细胞核RIP方案可能最适合发生在细胞核中的相互作用。如果尚无关于RNA结合位置的知识,则建议使用常规全细胞裂解物天然RIP方案(图2A)(17-700, 17-701),以提高结合RNA的免疫沉淀的可能性,不受结合位置的影响。该方案适用于各种数量和类型的起始材料,例如细胞和组织样品。如果已知结合RNA出现在细胞核中或与染色质相关,则建议使用细胞核天然(17-10522, 17-10523)或交联RIP(17-10520, 17-10521方案。这些方案不是从全细胞裂解物中免疫沉淀RNA,而是对分离的细胞核进行免疫沉淀,从而提高了信噪比。
尽管这些方案适用于所有类型RNA的免疫沉淀,但还有两种其他专门开发的方案可用于鉴定和分析与特定染色质相关长非编码RNA相互作用的染色质区域/RNA纯化染色质分离(ChIRP)(图2B) (17-10494, 17-10495)或m6A(N6甲基腺苷)RNA甲基化位点的全转录组谱图分析(图2C)(17-10499)。
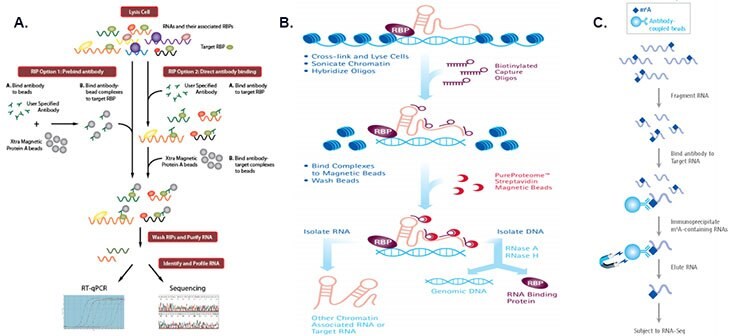
图 2.不同RIP方案的工作流程A. 全细胞裂解物的天然RIP工作流程(货号 17-700、17-701) B. ChIRP 的工作流程(货号 17-10494、17-10495) C. m6A RIP 的工作流程(货号 17-10499)
RIP(检测方案工作流程)下游分析
完成RIP方案后分离出的RNA可以通过几种分子检测方法进行分析,包括终点RT-PCR和定量RT-PCR(如果已知RBP的结合靶标)、微阵列或深度测序方法(图3)。根据具有已知序列的RNA靶标,可以设计基因特异性引物,以验证(和定量)免疫沉淀的RNA。确认RIP成功后,可以通过基于群体的方法(如针对所得cDNA的比较微阵列杂交)或通过对RIP的分子适应产物进行深度测序,对免疫沉淀中的RNA群体进行进一步分析。下面介绍了RIP实验的终点或实时定量测量或NGS分析的方法实例。
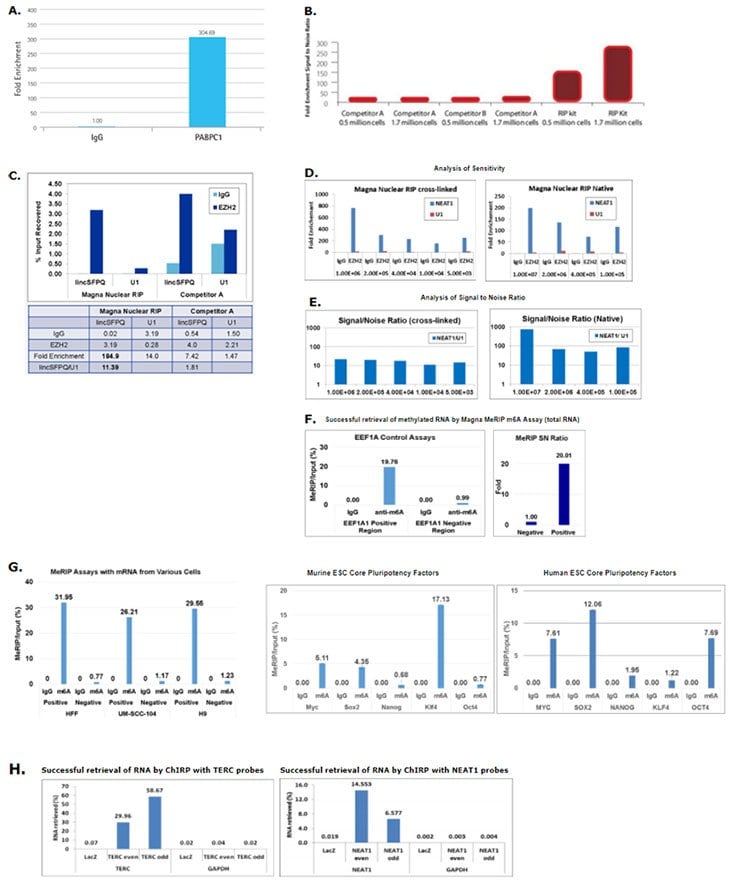
图 3.通过 qRT-PCR 进行 RIP RNAs 分析
A. 用5 µg正常小鼠IgG或PABPC1抗体和Magna RIP试剂盒(货号17-700)对HeLa细胞的RIP裂解物2x107细胞当量/IP)进行免疫沉淀。使用RIP引物ACTB,通过qRT-PCR方法验证了PABPC1相关RNA的成功免疫沉淀。
B.用2.5 μg正常兔IgG或HUR抗体和Imprint RNA免疫沉淀试剂盒(RIP)对HeLa细胞的RIP裂解物(0.5 × 106 或 1.7 × 106细胞当量/IP)进行免疫沉淀。使用对照引物Actin B和JUN,通过qPCR方法验证了HUR-靶标和非靶标相关RNA的免疫沉淀。
C. 利用qRT-PCR与Magna细胞核交联RIP(货号17-10520)分析HeLa细胞中与PRC2复合物(EZH2和SUZ12)和U1snRNA(阴性)相关的LincSFPQ。与竞争品牌试剂盒的信噪比性能进行比较。
图D和E. Magna细胞核RIP检测,分别使用低至5,000(交联,货号17-10520)和100,000(天然,货号17-10522)个细胞。灵敏度数据表面,使用5,000个HeLa细胞显示了交联方案NEAT1富集,而使用天然方法则需要100,000个细胞(C)。信噪比数据显示,天然方案的总体阳性与阴性比值更高(D)。
F. 使用来自HEK293细胞的总RNA进行MeRIP(货号17-10499)分析。然后,使用阳性对照引物(MeRIP引物人EEF1A1阳性,货号CS220017)和阴性对照引物(MeRIP引物人EEF1A1阴性,货号CS220018)通过RT-qPCR方法来分析纯化的RNA。
G. 左图:利用MeRIPTMm6A检测法和来自各种细胞的mRNA成功检测甲基化RNA。使用来自无外源性成分的人包皮成纤维细胞(HFF,货号 SCC058))、UM-SCC-104人头颈部鳞癌细胞(货号:SCC072)和人胚胎干细胞(H9)的mRNA,进行MeRIP(货号17-10499)。然后,使用阳性对照引物(MeRIP引物人EEF1A1阳性,货号CS220017)和阴性对照引物(MeRIP引物人EEF1A1阴性,货号CS220018)通过RT-qPCR方法来分析纯化的RNA。中图和右图:分析小鼠和人ES细胞中重编程基因的m6A RNA水平。使用来自PluriStem 129/S6 鼠 ES 细胞(货号SCR012)和H9 人 ES细胞的 mRNA,进行MeRIP(货号17-10499)。然后,使用Myc、Sox2、Nanog、Klf4和Oct4的MeRIP m6A 峰引物,通过RT-qPCR方法对纯化的RNA进行分析。小鼠重编程基因的PCR引物序列由Howard Chang(斯坦福大学)提供。人PCR引物是根据已发表的MeRIP-Seq数据设计的。使用Bio-Rad qPCR仪和iTaq ™通用一步法试剂盒(Bio-Rad)进行一步法RT-qPCR。通过利用0.1%输入样品或ΔΔCt方法绘制的标准曲线,计算输入回收率。
H 使用HeLa细胞裂解液及Magna ChIRP TERC lncRNA探针组ODD(偶数/奇数)(货号03-309)或NEAT1 lncRNA探针组ODD(偶数/奇数)(货号03-308)或Magna ChIRP™阴性对照探针组(LacZ) (货号03-307)进行 ChIRP(货号17-10494)。然后,使用RNA阳性对照引物(TERC或NEAT1)和RNA阴性对照引物(GAPDH),通过qRT-PCR方法对纯化的RNA进行分析。
RNA免疫共沉淀结合高通量测序技术(RIP-seq)
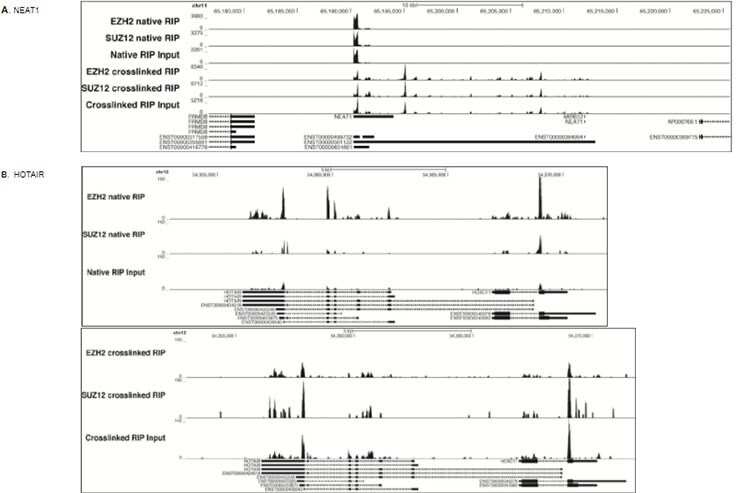
图 4.使用Magna细胞核天然RIP试剂盒(货号17-10522)或Magna细胞核交联RIP试剂盒(货号 17-10520)对HeLa细胞的EZH2和SUZ12进行 RIP-Seq 分析。数据显示,NEAT1(A.上图)和HOTAIR lincRNA转录子(B.中图和下图)处的信号丰富。
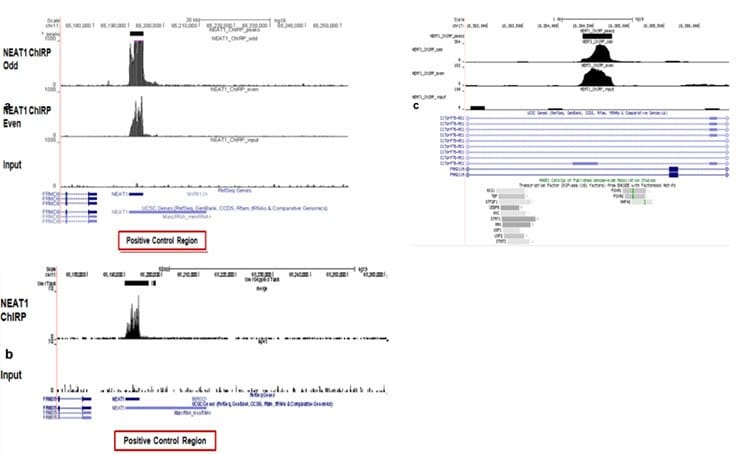
图 5.使用Magna ChIRP 试剂盒对DNA pull-down进行下一代测序分析。
利用HeLa细胞裂解物进行ChIRP。使用生物素化捕获寡核苷酸(Magna ChIRP™ NEAT1 lncRNA 探针组(odd组和even组)(货号03-308)和和阴性对照探针(Magna ChIRP™ 阴性对照探针组(LacZ) (货号03-307)) 进行pull down反应。在HiSeq仪器(Illumina)上对序列文库进行测序。使用Bowtie将序列读数与参考基因组(hg19)进行比对。(a) 分别使用偶数和奇数探针组的数据检出峰。MACS等算法可用于此用途。那些共有的峰被认为是有效的峰。(b)使用Howard Chang实验室的分析软件(https://changlab.stanford.edu/protocols.html)进行一系列比对后处理和过滤步骤。数据显示了NEAT1 RNA在NEAT1基因区域和另一个示例区域中的位置(c)。
RIP常见问题解答、故障排除技巧和数据分析指南
在开展RIP实验时,您可能会遇到一些困难,例如无RNA免疫沉淀、背景高或信噪比低。以下信息可帮助您在开始使用此技术前对其有充分的理解;在实验遇到困难时提供故障排除技巧并指导您进行数据分析。
- 全细胞裂解物RIP常见问题解答
- 全细胞裂解物RIP的故障排除技巧
- RNA纯化染色质分离(ChIRP)的故障排除技巧
- MeRIP的故障排除技巧
- RIP-qPCR简要方案
- RIP-qRT-PCR: 数据分析计算器
哪款RIP试剂盒适合您?
全细胞裂解物RIP
细胞核 RIP (交联和天然)
RNA纯化染色质分离(ChIRP)基础试剂盒和不同的 lncRNA 探针组
meRIP
如要继续阅读,请登录或创建帐户。
暂无帐户?