植物CRISPR/Cas9基因组编辑
ZFN诱导的基因靶向技术早在2003年就已发表。从那时起,靶向基因组编辑技术得到了迅速发展,并已商业化。最近,CRISPR/Cas9 通路的发现加快了对该领域的兴趣,为研发开辟了新的可能性。虽然 CRISPR 通路在细菌中被鉴定为假定的适应性免疫系统的一部分,但它很快适应了修饰真核生物基因组的目的。虽然像 ZFN 这样的工具为今天的基因组编辑奠定了基础,但这种创始技术和其他类似的技术存在局限性:蛋白质:ZFN的DNA 相互作用使得其设计变得复杂,ZFN 表达构建体的组装非常耗时,并且 ZFN 靶向的选择在许多富含 A-T 的植物基因组中受到限制。CRISPR通路已经被采用和修改用于真核基因组编辑,克服了许多障碍:它依赖于RNA:DNA相互结合作用以找到其基因组靶标,该结合体的识别序列是易于改变的18-20个碱基对,并且核酸酶结合的唯一要求是在靶位点旁边存在NGG。CRISPR/Cas9 在研究使用农杆菌瞬时表达分析的植物中的首次于 2013 年报道。CRISPR/Cas9 技术已成功应用于模式植物(本氏烟草、拟南芥)和作物(水稻、小麦),且名单正在不断增加。
- CRISPR/Cas9:它是什么?它是如何工作的?
- 利用 CRISPR/Cas9 进行基因组编辑的优势
- 简化工作流程:植物中的 CRISPR/Cas9
- 用于单子叶植物和双子叶植物的即用型 Cas9 和向导 RNA (gRNA) 表达质粒
- 自定义 CRISPR/Cas9 植物产品
CRISPR/Cas 9:它是什么?它是如何工作的?
CRISPR代表"Clustered Regularly Interspaced Short Palindromic Repeats"(成簇规律间隔短回文重复序列)。II型原核生物CRISPR“免疫系统”的发现,使得能够开发简单、易用、快速实施的RNA引导的基因组编辑工具。CRISPR/Cas9 系统由单个单体蛋白和嵌合 RNA 组成。gRNA中的20-nt序列具有序列特异性,并且裂解是由Cas9蛋白介导的。沃森-克里克碱基配对与靶 DNA 序列是基于 gRNA 的裂解的基础,使得每个靶标的复杂蛋白质工程变得不必要。仅需要修改gRNA中的20nt以便于识别不同的靶标。
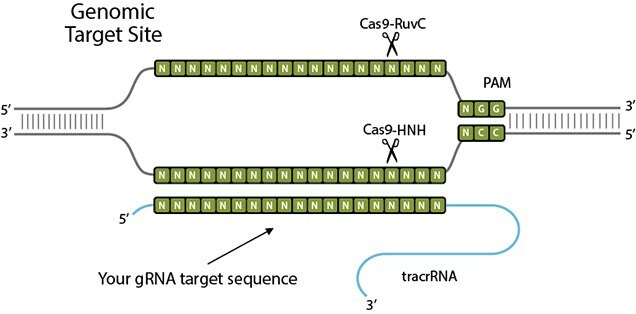
CRISPR/Cas9 由 Cas9 蛋白、CRISPR RNA (crRNA) 和反式激活 crRNA (tracrRNA) 组成。在基因编辑应用中,crRNA和tracrRNA通常融合成单个向导RNA(sgRNA)。核糖核蛋白以 crRNA 引导序列侵入靶标,形成 20-bp 的 RNA/DNA 杂交体,遇到原型间隔区相邻基序 (PAM) 后置换相对的 DNA 链,如 NGG。Cas9内切核酸酶随后用HNH核酸酶结构域切割互补DNA链(靶链),并用RuvC样核酸酶结构域切割置换的DNA链(非靶链)以产生双链断裂(DSB)。宿主细胞通过非同源末端连接(NHEJ)或同源定向修复(HDR)途径修复DSB可用于产生基因敲除或通过与DNA供体的同源重组引入特定的基因修饰。
与其他基因编辑方法相比,由化脓性链球菌来源的 Cas9 蛋白和向导 RNA (gRNA) 组成的 RNA 引导核酸内切酶 (RGENs) 可以通过仅替换RNA组分来定制,导致与其他基因编辑方法相比减少劳动和时间。通过使用根癌农杆菌或转染编码它们的质粒,可编辑的核酸酶可以被输送到植物细胞中,其中这些核酸酶以序列依赖性方式切割染色体靶位点。 结果是位点特异性DNA双链断裂(DSB),其通过内源系统修复导致靶向基因组修饰。
利用 CRISPR/Cas9 进行基因组编辑的优势
CRISPR/Cas9 的主要优势在于简单性、可用性、节约成本和多功能性。
CRISPR/Cas9 系统不需要任何蛋白质工程步骤,使得测试每个靶基因的多个 gRNA 更加直接。
gRNA 序列中仅20个核苷酸具有不同的靶特异性,这意味着克隆也是不必要的。
通过使用两个互补的退火寡核苷酸进行体外转录,可以产生任意数量的gRNA。这使得大型 gRNA 文库的组装成本低廉,因此 CRISPR/Cas9 系统可用于高通量功能基因组学应用。
与 ZFNs 和 TALENs 相比,CRISPR/Cas9 的另一个优势是可重复使用。在多个位点同时引入 DSB 可用于同时编辑多个基因。 敲除冗余基因或平行途径可能特别有用。研究人员可以通过靶向同一染色体上两个间隔较宽的切割位点来设计大的基因组缺失或倒位。使用 CRISPR/Cas9 系统进行多重编辑只需要单体 Cas9 蛋白和任意数量的不同序列特异性 gRNA。相反,使用ZFN或TALEN进行多重编辑需要每个靶位点特异的单独的二聚体蛋白质。
CRISPR / Cas9系统可以切割人类细胞中的甲基化DNA,从而允许基因组修饰超出其他核酸酶的范围。虽然这尚未在植物中进行特异性研究,但有理由相信切割甲基化DNA的能力是CRISPR / Cas9系统固有的并且不依赖于靶基因组。
简化工作流程:植物中的 CRISPR/Cas9
随着基因组编辑技术的引入,植物生物技术正在进入一个新时代,这些技术能够精确操纵特定的基因组,从而取代了较旧的随机诱变方法,如EMS诱变和g-辐射序列。植物CRISPR/Cas9产品用于农杆菌介导的植物转化或基因枪微粒轰击或原生质体转化。该产品基于衍生自化脓性 链球菌的IIA型CRISPR/Cas9。对天然 Cas9 编码序列进行密码子优化,分别在单子叶植物和双子叶植物中表达。单子叶植物 Cas9 构建体含有用于 sgRNA 表达的单子叶植物 U6 启动子,双子叶植物 Cas9 构建体含有双子叶植物 U6 启动子。植物选择标记包括潮霉素 B 抗性基因、新霉素磷酸转移酶基因和bar基因(膦丝菌素乙酰转移酶)。
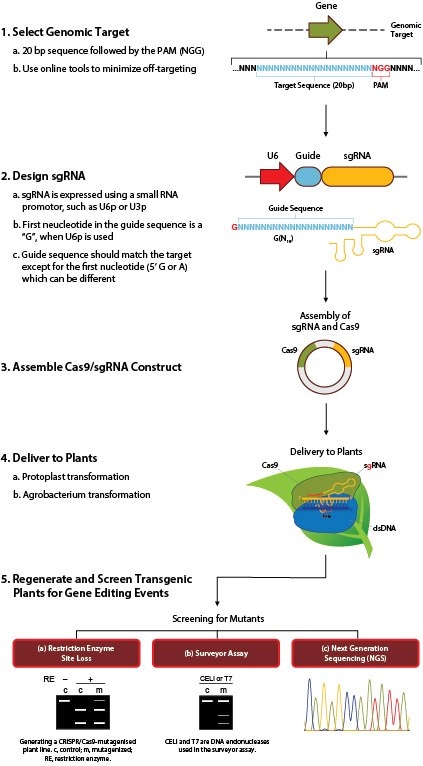
产生CRISPR / Cas9诱变植物系的途径。 c,控制;m,诱变;RE,限制酶。CELI和T7E1是测量员测定中使用的DNA核酸内切酶。
用于单子叶植物和双子叶植物的即用型 Cas9 和向导 RNA (gRNA) 表达质粒
- 我们的植物CRISPR / Cas9产品用于农杆菌介导的植物转化或基因枪微粒轰击或原生质体转化。
- 密码子优化的Cas9蛋白和gRNA由单一载体表达,并以即用型转染级DNA的形式进行提供。
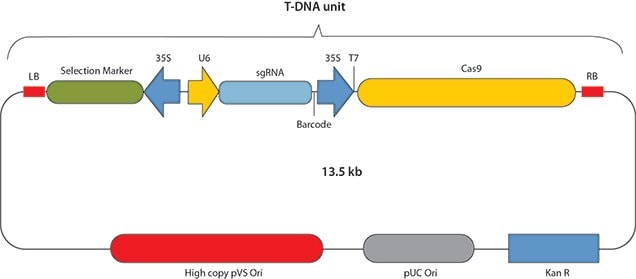
用于农杆菌介导的转化的CRISPR/Cas9构建体的基本结构。
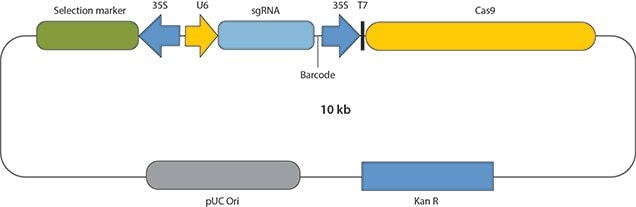
用于基因枪或原生质体转化的 CRISPR/Cas9 构建体的基本结构。
CRISPR植物对照
参考文献
如要继续阅读,请登录或创建帐户。
暂无帐户?