CRISPR / Cas核酸酶RNA介导的基因组编辑
CRISPR/Cas是什么?
CRISPR代表"Clustered Regularly Interspaced Short Palindromic Repeats"(成簇规律间隔短回文重复序列)。II型原核生物CRISPR“免疫系统”的发现,使得能够开发简单、易用、快速实施的RNA指导的基因组编辑工具。CRISPR首字母缩写通常发音为“ crisper”。
CRISPR / Cas采用何种工作原理?
CRISPR通路在细菌中被发现,其功能很像免疫系统,可以抵御入侵的病毒和质粒DNA。来自入侵病毒的短DNA序列(间隔区)掺入细菌基因组内的CRISPR基因位点,并充当先前感染的“记忆”。再感染引发互补的成熟CRISPR RNA(crRNA)以找到匹配序列 — 其为CRISPR相关(Cas)核酸酶提供特异性,以在特定 “外源” DNA序列处形成双链断裂。
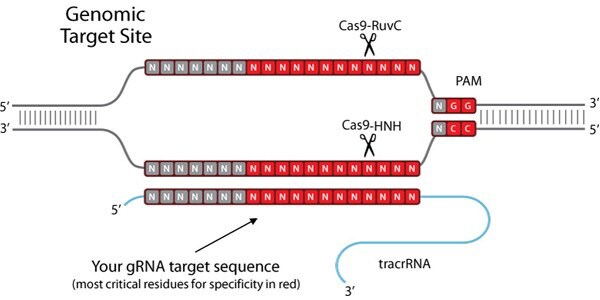
图 1.基因组靶标位点
CRISPR Cas9采用何种工作原理?
为了创建这样的工具,内源CRISPR途径被简化为两个主要组分:Cas9核酸酶和指导RNA(gRNA)1-7。导向RNA是由crRNA和tracrRNA组成的双组分系统。crRNA靶向待切割的双链DNA,并且具有短的同源区域,允许其结合tracrRNA。 tracrRNA提供与Cas9蛋白结合的茎环结构。crRNA:tracrRNA双链体被称为gRNA。 Cas9核酸酶和gRNA形成Cas9核糖核蛋白(RNP),可以在整个基因组环境中结合并切割特定的DNA靶标。为了被RNP切割,靶必须具有两个特定序列。首先,gRNA需要17-21个碱基的RNA至DNA同源性,这被称为前间隔序列。 其次,Cas9蛋白需要有一个短的前间隔序列邻近基序(PAM),以结合靶DNA(参见图2)。 如果存在连接tracrRNA,并且在gRNA和基因组靶标之间存在足够的同源性,则RNP切割靶DNA的两条链,在基因组中的该精确位置处产生DSB。
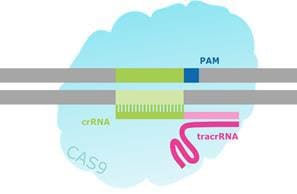
图 2.CRISPR / Cas靶向双链断裂的示意图。
虽然细胞核中的功能性CRISPR是RNP形式,但作为分子工具的CRISPR的组件适合于各种递送方法。 早期实验成功地创建了嵌合单指导RNA或sgRNA,其将crRNA和tracrRNA组合成单个RNA链而不是天然存在的双链体。 该sgRNA和Cas9 mRNA可以从单个质粒表达,用于直接转染或包装成用于慢病毒转导的颗粒。 也可将重组Cas9蛋白与合成产生的crRNA和tracrRNA组合,以生成RNP转染或显微注射给胚胎。
靶向DSB形成后,细胞通常使用两种DNA修复途径中的一种来存活:非同源末端连接(NHEJ)或同源依赖性修复(HDR)。这些修复机制经常会出错,导致在靶位置诱变,或者通过破坏编码序列来功能性地失活或敲除基因(NHEJ),或者通过添加新的DNA序列来敲入特定的序列变化(HDR)。通过这种方式,CRISPR / Cas9系统能够对染色体DNA进行永久的、可遗传的修饰。此外,当Cas9核酸内切酶结构域失活并附着于其他效应分子以作用于基因组中的gRNA指定位点时,CRISPR系统可用作靶向递送系统。 这将CRISPR系统功能扩展到基因激活和抑制。最后,CRISPR系统可用于筛选。CRISPR / Cas9系统的基因敲除、基因敲入、基因激活或抑制、以及筛选这所有四种主要用途将在下面进一步讨论。
基因敲除
当与靶向基因特异的gRNA一起共表达时,CRISPR / Cas9产生敲除细胞或动物模型。基因敲除的目的是通过破坏其在细胞中的表达来揭示基因功能。共表达的适当设计的gRNA指导Cas9切割靶序列,并在目的基因中产生DSB。然后可以通过三种方式中的任何一种实现基因敲除:1)细胞通过NHEJ修复断裂,导致切割基因的开放阅读框(ORF)内的随机插入或缺失(“插入缺失”);2)细胞通过HDR从用户提供的模板修复断裂,将特定的破坏性序列插入ORF中;3)一对gRNA产生两个DSB,其位于基本编码序列的侧翼,导致其切除。
NHEJ是最活跃的修复机制,但是容易出错,并且经常引入移码,导致过早终止密码子和/或导致所得转录物成为无义介导的衰变(NMD)的靶标。移码修饰可导致过早终止密码子被引入转录物,阻止氨基酸链的关键部分被翻译,导致异常短的无功能蛋白质。无义介导的衰变通过消除含有过早终止密码子的mRNA转录物来减少基因表达中的错误。 理论上,当外显子-外显子连接复合物(也就是RNA剪接期间在外显子之间的前mRNA链上形成的蛋白质复合物)在转录物被剪接后未被核糖体适当地去除时,这个监视途径被激活。当外显子-外显子连接复合物由于上游移码而保持结合时,转录物被指示降解,并且功能蛋白质从未被翻译。这两种途径都有效破坏细胞中的基因功能。
基因敲入
CRISPR / Cas9也可用于引入或“敲入”新的DNA序列。常见的修饰包括引入单核苷酸多态性(SNP)、小标签、loxP或更大的基因盒,例如荧光蛋白。这些修饰是通过特定位置的Cas9诱导的DSB进行的,这显著增强了靶向整合的机会。靶向整合(基因敲入)通过HDR发生。为了通过HDR进行基因编辑,必须将含有所需序列的DNA “供体” 或修复模板与gRNA和Cas9一起递送至细胞,通常在供体质粒或寡核苷酸上。基因敲入的效率通常低于敲除(<10%的修饰等位基因),但可用于产生范围从单核苷酸变化至大插入物的特定修饰。
基因激活和抑制
除了作为基因组编辑工具之外,CRISPR还可以用作其他功能蛋白的靶向递送系统。Cas9的一个独特特征是它能够独立于DNA切割而结合靶DNA,因为这是Cas9机制的两个独立步骤。野生型Cas9具有两个核酸酶结构域:RuvC和HNH。为了在没有切割的情况下实现结合,通过诱导点突变(SpCas9中的D10A和H840A)使两个核酸酶结构域失活,导致核酸酶死亡的Cas9(dCas9)。当与靶向转录起始位点的gRNA组合时,发现单独的dCas9足以通过阻断转录起始来降低或抑制转录。在这一发展之后,科学家开始尝试将转录抑制因子和激活因子与dCas9联系起来。KRAB转录抑制结构域已经与dCas9融合,作为效应结构域介导的转录沉默的模式。dCas9也与转录激活因子和转录效应蛋白融合。用于激活的dCas9是广泛流行的研究领域,并且包括诸如dCas9-VP64、dCas9_SunTag、dCas9-SAM、dCas9-RNA支架和dCas9-VPR的系统。 在我们的产品线中,我们将dCas9与人E1A相关蛋白P300的催化组蛋白乙酰转移酶(HAT)核心结构域融合。当与靶向转录起始位点和增强子区域的gRNA组合时,dCas9-P300指导组蛋白乙酰化,从其异染色状态释放DNA,以通过天然染色体环境中的内源细胞机制上调基因表达。dCas9-P300组蛋白乙酰化方法代表了相对于dCas9-VP64或其他类似基因活化基序的独特作用机制。
筛选
筛选可以快速调查大量候选基因或基因组位点,以参与您的途径或感兴趣的表型。池化寡核苷酸生产和大数据处理的改进,加上慢病毒递送的效率,使研究人员能够同时考虑数千个候选基因,无论是作为文库还是更为集中的子集(panel)。我们将文库定义为CRISPR克隆的全部基因组集合;而子集(panel)则是较小的基因子集。通常,子集(panel)和文库可以以两种形式组装:池化(在一个管中有数千个gRNA)或排列(在96孔板中每个孔一个gRNA)。这两种方法都能快速地为更大的问题提供答案,与过去的方法相比具有明显的优势,过去的老方法需要一次一个地询问候选基因,是一个费时费力的过程。
我们提供多种筛选解决方案,包括来自Broad的池化GeCKO文库,以及Sanger阵列全基因组慢病毒CRISPR文库,以执行大型基因敲除筛选(8)。并且,人类和小鼠CRISPR / Cas9协同激活调控子(SAM)池化文库亦即将推出,用于全基因组筛选转录激活表型!
CRISPR / Cas9是什么?
CRISPR / Cas9系统于1993年被发现,并且显示在细菌和古细菌生物体内进化,作为对病毒和外来质粒的防御。天然CRISPR途径的功能是将核酸酶靶向侵入性DNA,产生潜在致命的双链断裂(DSB)。在识别该途径的功能后不久,来自细菌化脓性链球菌(SpCas9)的II型CRISPR / Cas9系统被重新利用以产生简单但强大的分子工具,其可被编程以将核酸酶靶向特定的基因组序列。
我们为开发CRISPR/Cas9所作的贡献
我们很荣幸能为全球研究圈提供最新的CRISPR基因组编辑工具。作为近十年来第一家商业化供应靶向基因组编辑技术的公司,我们这方面的专业知识业内难以匹敌。对基因组编辑工具有特定靶向性和稳定切割活性关键要求时,这一经验尤为重要。我们在全新CRISPR产品线兼具这两种特性,现可通过我们快速简单的网页设计平台轻松获取。我们的CRISPR产品可通过下方链接直接订购,也可浏览此页面上的CRISPR内容了解有关该技术的更多信息。
全功能即用型Cas9和指导RNA(gRNA)表达质粒
- 密码子优化的Cas9蛋白和gRNA从单一载体表达,并作为即
用型转染级DNA提供。 - 预先设计的独特CRISPR位点可最大限度地减少脱靶,可用于人类、小鼠和大鼠基因组的编码区域。可应要求随时提供任何其他区域或物种的定制设计(参见定制CRISPR索取表)。
- GFP或RFP与2肽融合到Cas9的C末端,通过FAC分选跟踪转染效率和丰富基因组编辑活动。
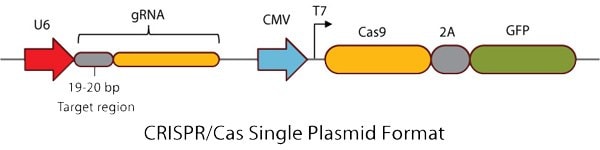
图 3.CRISPR/Cas单质粒形式
CRISPR配对切口酶
- CRISPR配对切口酶通过要求将两个单独的gRNA独立结合到局部基因组区域,进一步最小化脱靶双链断裂。
- 在存在Cas9-D10A切口核酸酶的情况下,两个gRNA在DNA的相反链上诱导单链断裂,产生功能性双链断裂。
- 预先设计的、独特的CRISPR配对切口酶可用于人类、小鼠和大鼠基因组的编码区域(配对切口酶预设计)。可应客户要求随时提供任何其他区域或物种的设计(参见定制CRISPR索取表)。
- 当为特定靶订购Sigma的CRISPR配对切口酶时,研究人员将获得两个单独的即用型转染级质粒,表达来自人U6启动子的gRNA。Cas9-D10A切口核酸酶必须作为质粒或mRNA单另购买。
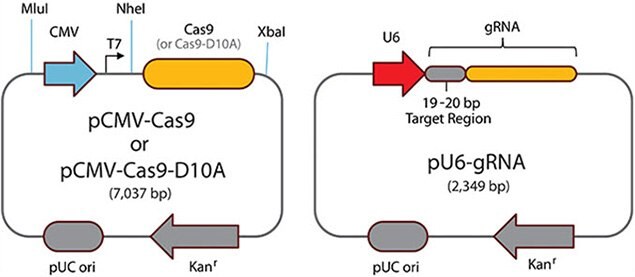
图 4.配对Cas9切口酶

图 5.CRISPR Cas3
配对Cas9切口酶 — 为获得最佳Cas9-D10A配对切口酶功能,指导RNA应设计为5'至5'方向,PAM间距为30至150 bp。
仅含纯化RNA的导向RNA
- 适用于显微注射或细胞培养的纯化RNA形式的即用型gRNA。
- 使用与Sigma CRISPR和CRISPR配对切口酶质粒相同的严格规则所设计。
- CRISPR RNA格式是创建转基因动物模型的研究人员或关注基因组质粒整合的研究人员的理想选择。
- RNA形式避免了对特定启动子的需要,允许在大多数已知细胞类型和生物体中表达。
- 野生型Cas9(CAS9P)或Cas9-D10A切口(CAS9D10AP)核酸酶必须作为质粒或mRNA(CAS9MRNA,CAS9D10AMRNA)单另购买。
- 可提供供体设计。

图 6.慢病毒CRISPR:(A) 全功能慢病毒CRISPR载体的载体图。(B) 用通过pLV-U6g-EPCG制成的颗粒转导HEK293细胞后的GFP信号。(C) 用慢病毒(pLV-U6g-EPCG)转导HeLa细胞,然后在嘌呤霉素和6TG处理上进行选择以富集HPRT1敲除。通过错配测定(CEL-I)在gRNA靶位点检测到插入和缺失。
CRISPR阳性和阴性对照
- 经过测试和验证的人EMX1基因阳性对照提供了参考基础,并允许研究人员对其基因组编辑实验的效率进行并行评估。
- 可提供即用型人单个gRNA EMX1阳性对照(CRISPR01-1SET)和CRISPR配对切口酶(CRISPR02-1SET)。
- 每个人EMX1阳性对照都带有野生型Cas9 (CRISPR01-1SET) 或切口酶Cas9-D10A核酸酶(CRISPR02-1SET)的等分试样。
- CRISPR通用阴性对照(CRISPR06-1EA、CRISPR07-1EA、CRISPR08-1EA)是全功能即用型转染级质粒,在CMV启动子下表达野生型Cas9核酸酶,以及设计用于靶向未知的人、小鼠或大鼠基因的gRNA序列。

图 7.CRISPR01阳性对照(标为EMX1s4+Cas9的泳道)以及CRISPR02阳性对照(标为EMX1s4, EMX1as4+D10A的泳道)的凝胶 I 图像
产品列表
参考文献
如要继续阅读,请登录或创建帐户。
暂无帐户?