锁核酸
LNA是一种新型核酸类似物,含有2'-O,4'-C亚甲基桥(图1)。这种桥接锁定在3'-内构象中,限制了呋喃核糖环的灵活性,并将结构锁定为刚性双环形式。这意味着更强的分析性能和更广泛的应用。
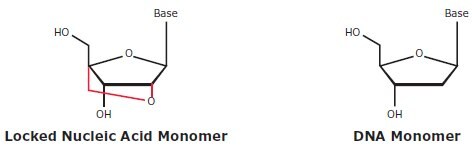
图 1.LNA和天然状态DNA单体的结构。
PCR引物、qPCR探针和其他类型寡核苷酸中的锁核酸可溶于水和标准缓冲液,并遵循Watson-Crick碱基配对规则。1
优点
当LNA被掺入qPCR探针时,与天然状态DNA碱基相比,它具有多种优势,包括:
- 热稳定性和杂交特异性更高
- 基因定量和等位基因鉴别更准确
- 针对有问题的靶序列,探针设计更容易、更灵活
热稳定性和杂交特异性更高
将LNA掺入qPCR探针可提高双链体热稳定性2,并提高探针与靶序列杂交的特异性。3 这减少了来自伪结合的背景荧光,从而提高了信噪比。此外,与天然状态DNA qPCR探针相比,LNA探针与其靶标的杂交得到改善,在培养基盐条件下,每取代一个LNA单体,解链温度 (Tm)提高多达8℃4(表1)。杂交的这种增加大大拓宽了测定条件范围,实现更成功的多重测定。5
基因定量和等位基因鉴别更准确
掺入LNA碱基后,qPCR探针通过SNP区分等位基因的能力大大增强6-8(图2)。与天然状态DNA qPCR探针相比,单碱基错配的存在对LNA qPCR探针与其靶标之间的双链体形成具有更大的去稳定作用。掺入LNA碱基的较短qPCR探针可以在与较长天然状态DNA qPCR探针相同的温度下使用。
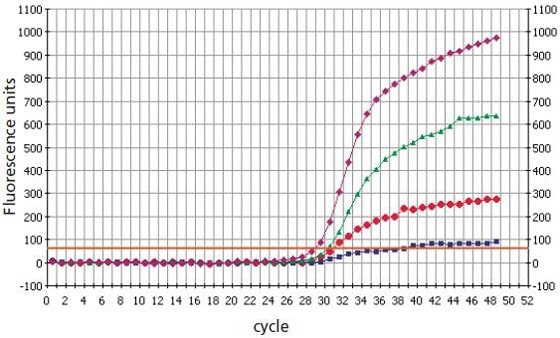
图 2.LNA双标记探针在SNP基因分型分析中比DNA双标记探针具有更好区分能力9。粉色)用LNA突变探针(具有3个LNA碱基的16聚体)进行突变DNA分析。绿色)用天然状态DNA突变探针(25聚体)进行突变DNA分析。红色)具有天然状态DNA突变探针(25聚体)的野生型DNA。紫色)具有LNA突变探针(具有3个LNA碱基的16聚体)的野生型DNA。
针对有问题的靶序列,探针设计更容易、更灵活
由于LNA的杂交特征改善,并伴随着Tm增加,LNA qPCR探针可以被合成得更短,这克服了天然状态DNA qPCR探针存在的某些设计限制。具体而言,可以设计LNA qPCR探针来解决传统上有问题的靶序列,例如富含AT或GC的区域。例如,富含AT的天然状态DNA qPCR探针通常需要超过30个碱基(有时超过40个碱基)以满足扩增子设计指南,但仍可能表现不佳。使用LNA qPCR探针,LNA碱基的选择性放置有助于优化设计高度特异性、较短的qPCR探针,即使仅有13至20个碱基,也能表现良好。此外,LNA极大地促进了用于靶向困难SNP的qPCR探针的设计,例如相对稳定的G:T错配。
其他优点
LNA qPCR探针与所有实时热循环仪和终点分析检测仪器兼容。不需要专门的工具。
应用
LNA可以掺入所有可用的qPCR检测化学品,包括:
- SYBR® Green 引物
- 双标记探针
- Molecular Beacons
- LightCycler® 探针
- Scorpions® 探针
并且,可用于以下应用:
- SNP检测
- 等位基因鉴别
- 病原体检测
- 多重测定(Multiplexing)
- 病毒含量定量
- 基因表达分析
- 基因拷贝测定
其还可用于以下序列:
- 反义寡核苷酸
- 脱氧寡核苷酸
- 捕获探针
- 核酸适配体
- 核酶
参考文献
如要继续阅读,请登录或创建帐户。
暂无帐户?