PCR/qPCR/dPCR实验设计
整个PCR工作流程易受引入变量的影响。许多变量无法避免,例如样本来源和逆转录步骤的要求。实验设计也波动较大,可能决定PCR的成败,影响实验的可重复性和灵敏度。实验设计过程遵循以下逻辑流程:第一步是确定靶标的位置。在某些情况下,寡聚核苷酸序列是由特定应用决定,且不可避免,例如SNP检测;另一些情况下,可以使用整个基因,例如拷贝数测定。一旦选择了大概的实验位点,就能确定最合适的引物并确定修饰。进行多重PCR实验时,重点要考虑反应中的寡核苷酸之间可能发生的相互作用,以及靶标的相对丰度。在复杂情况下,例如目的是检测非常低的拷贝数或靶标浓度的微小差异,建议选择并测试几种引物组合,然后结合适合的探针组合。
采用合适的设计软件能极大地促进实验设计的进程。OligoArchitect提供了两种设计供选择。首先是OligoArchitect Online,是一种具有多种功能的软件设计工具。如果实验设计需要专门的功能,第二种选择就是OligoArchitect Consultative软件,包含我们专业的分子生物学家协助。
扩增子选择
扩增子是待分析的靶标序列,包含在正向和反向PCR引物之间。扩增子大小的确定部分取决于所使用的分析方法。当通过凝胶电泳可视化PCR片段时,需要PCR片段足够长,使得DNA结合染料能有效染色,并与所选的人工长度标记的范围相适应。同样地,当通过毛细管电泳仪分析片段时,PCR产物将介于两者之间,100个碱基对和超过2kb的任何位置(最终被酶反应限制)。
当使用qPCR进行最终读数时,选择较小的扩增子以确保在每个循环中进行准确定量。理想情况下,qPCR扩增子的长度范围为75至200个碱基,除非设计限制使得引物超过该范围。事实上,片段大小可以通过考虑一些因素来确定,包括生物学考虑。
检测位置可根据实验的目的事先确定:理想情况下,用于确定mRNA是否存在或含量的检测定位于外显子-外显子连接处,以避免检测到污染的gDNA序列。然而,这些区域通常高折叠;因此,需要做一个决定,是优先选择一个可能性能较差的外显子跨越实验,还是一个更高质量的外显子实验。相比来自多拷贝基因的低丰度转录物,如果mRNA丰度高且从单拷贝基因转录而来,gDNA污染信号的影响要小一些。SNP的检测需要在错配位点上定位探针或3'端引物。
剪接体的分析需要根据靶标专门设计方法。有时候需要同时检测所有的剪接体,这可以通过选择在所有变体之间保守的外显子区域来实现。然而,研究不同剪接体之间的表达差异则需要更有创造力的方法。此次示范实验目的是检测图6.1中所示的哪些替代转录物被表达了。由于外显子相对较小,扩增所有外显子产物大约为300个碱基,比扩增剪接体的产物长度短一些。本研究所选择的设计为:
- 在每个外显子连接处设计几个实验,并在每个实验中探测每种样品。
- 设计外显子1的3'端引物和外显子4的5'端引物。扩增任何由外显子组合组成的转录物,都会产生特定长度的扩增子。转录的定义将用qPCR确认及SYBR Green I染料反应熔解曲线确定。

图 6.1.四种潜在剪接体的表达示意图。区分每种剪接体时,可通过跨越每个外显子连接的特定引物对设计,通过外显子1和外显子4的通用引物对的扩增,以及使用qPCR、SYBR Green I染料熔解分析方法。
一般通过以下标准评估扩增子序列:
- 建议对靶标序列区域进行初步评估。
- 确认没有异常SNP。引物和模板之间的一个错配会使得熔解温度降低10°C,从而影响PCR的效率。
- 确认所选序列与目标物种的基因组/转录组中任何其它序列不具有同源性。靶向多种器官系统时(例如病原体检测),必须对样品中所有的序列进行同源性检测。
- 使用OligoArchitect的折叠算法检测靶序列可能的二级结构,使用选定的设计软件或mfold软件(http://mfold.rna.albany.edu/)模拟在设计的引物退火温度下模板的折叠。选择预测为开放结构的模板区域,避免非常负ÄG值的茎环二级结构。当使用一步法发转录方法和基因特异性引物时,这点需要重点考虑。
- 避免回文序列和重复区域。
- 避免G/C含量高的区域,目标是50%左右的GC含量。
- 设计多重实验时,扩增子应尽可能具有相似的长度和GC含量,避免偏倚扩增。
- 设计针对基因家族的实验时,根据需要确定同源性或异质性区域。然后对序列进行比对,并检查合适的共有序列的片段。
- 对于转录物特异性设计(以避免检测到gDNA模板),尽可能在外显子-外显子连接区域以外选择靶向区域。
转录物特异性扩增子的选择
大多数(但非全部)DNA在RNA纯化过程中从样品中去除的。为避免在RT-qPCR过程中扩增DNA,建议选择的引物要么在一个大内含子的两侧(不在mRNA的序列中),要么跨过一个外显子-外显子连接(图6.2)。
EnsemblGenomes网站(http://ensemblgenomes.org/)提供了许多脊椎动物、细菌、原生生物、真菌、植物和无脊椎后生生物物种的已知基因的内含子/外显子注释。另外,如果靶标基因组和cDNA序列都可以公开获得,可以在目标生物的基因组数据库中,用BLAST搜索检索cDNA序列确定内含子的位置(图6.3)。图6.3中的内含子1太长(~6.5kb),DNA不该在常规PCR或受控PCR条件下扩增。但是,其它的内含子相对较短(<1 kb),因此RT-qPCR期间可能会扩增DNA(例如,参考实验优化和确认)。引物必须跨越外显子-外显子连接,或位于长内含子(几kb)的两侧,或在几个小内含子的两侧。

图 6.2.(A)跨越内含子和(B)侧翼连接内含子的RT-PCR引物示意图。内含子为红色,外显子为绿色。引物P1和P2跨越内含子,引物P3和P4位于内含子两侧。注意引物P1和P2不会产生PCR产物,除非退火温度极低。如果内含子较短(~1 kb),P3和P4可能会产生长于预期的PCR产物,但如果内含子足够长(几kb)则不会。

图 6.3.用BLAST将cDNA序列和基因组DNA序列进行比对。来自Genbank的大鼠p53的完整cDNA序列(登记号NM_030989)用megaBLAST在大鼠基因组(blastn)中搜索相同的序列。cDNA和gDNA的比对结果显示位于10号染色体上。通过该信息,cDNA的外显子可以与gDNA相应的区域对齐,引物可直接针对由长内含子分开的外显子设计,比如外显子1和外显子2。
甲基化特异性检测
DNA甲基化是细胞分化的重要部分,会导致基因表达以稳定的方式发生改变。甲基化对高等生物的正常发育很重要,并且可以遗传。DNA甲基化基因调控,包括将甲基加到胞嘧啶嘧啶环的5号位或腺嘌呤嘌呤环的6号氮上。在成人体细胞组织中,DNA甲基化通常发生在CpG二核苷酸环境中,而非CpG甲基化在胚胎干细胞中普遍存在。甲基化特异性实验需要鉴定序列内的CpG岛——通常位于基因的启动子区域。该信息在使用Beacon Designer(Premier Biosoft)时自动定位,可从以下网站获取:http://www.mybioinfo.info/index.php。
引物设计
本章中描述的一般引物和探针设计的建议对多种应用都适用,包括基因表达研究、SNP检测、甲基化检测研究、拷贝数测定、病毒载量检测和剪接体定量。每种应用也特定的设计考虑因素,接下来会单独讨论。
引物设计的一般标准
对于大多数应用,引物设计成与待扩增的模板DNA序列完全互补。PCR引物设计的基本考虑因素包括:
- qPCR引物长度通常为20-24个核苷酸,熔解温度(Tm)约为60°C
(59±2°C),但常规PCR可能有所不同,为(55±5°C)。特定应用可能需要修改引物长度和Tm。 - 引物的GC含量应为40-60%,并且不应有明显的二级结构。
- 引物自身及引物之间不应存在互补,特别是3’末端。这降低了扩增过程中产生引物二聚体产物的可能性。
- 避免3’端发夹结构(检查3’端5个碱基,3个为A或T,2个为G或C即可)。
- 避免运行长度超过4个重复相同核苷酸或回文区域。
SNP特异性引物
在某些情况下,例如在设计单核苷酸多态性(SNP)测定时,测定的位置不灵活,周围的序列也会影响到所选的寡核苷酸序列。认识到临床条件与种系和获得的体细胞SNP之间的关系,对开发更加敏感和特异的检测系统起到推动作用。这反映了使用寡核苷酸杂交区分SNP存在的难题。这些难题是由不同的错配之间不稳定的差异引起的。其中G:A,C:T和T:T可能具有很强的破坏稳定的作用,G:T和C:A比较弱,因为可以形成氢键,因此难以将这些配对与天然G:C和T:A区分开来。许多系统都是根据扩增-受阻突变系统(ARMS)1改编的,它已经被广泛应用,并有助于筛查囊性纤维化突变2。ARMS引物长30个碱基(较长的引物,最多60个碱基,是有功能的)。3’的碱基是SNP特异性的,因此对靶序列(正常或突变碱基)具有特异性。在倒数第二的位置引入一个额外的错配。这是考虑到邻近的碱基和SNP不匹配而确定的(表6.1,摘自Little,2001)。
经《Current Protocols in Human Genetics》允许转载:Little, S. 2001.Amplification-Refractory Mutation System (ARMS) Analysis of Point Mutations.Curr.Protoc.Hum.Genet.7:9.8.1–9.8.12.
其他研究小组使用了类似的观点并证明了引物中N-2和N-3位置引入错配的作用,Liu等人3对错配的相对位置进行了深入分析,以获得最大的不稳定作用,因此具有最高的特异性。
多重PCR
要在每管中增加PCR的通量或节约样品材料时,需要使用多重PCR同时扩增多个靶标。引物设计是多重PCR成功的关键因素。遵循引物设计的一般标准,并对包含在反应中的所有引物(和探针)进行相容性验证至关重要。在一些情况下,使用略长的、Tm约为65℃的引物可能是有利的。如果要根据大小不同来分析得到的扩增子,则须在实验设计时就考虑分析方法的分辨能力。试图用qPCR定量多个靶标时,扩增子应尽可能相似以避免扩增偏倚。除引物外,模板不能形成稳定的二级结构也十分关键,因为这会阻碍PCR的进行。如果我们已经知道靶标之间浓度差异很大,则在高浓度的靶标中引入阻断引物可能有利于促进准确地检测出低浓度靶标4。
非编码RNA定量
与编码基因组相反,估计约有97%的人转录组由非编码RNA(ncRNA)组成5;6,7。长的非编码RNA是该家族的一员,被描述为一类调节RNA分子。这些分子在表观遗传学,发育,癌症和关键的生物学过程中发挥作用8,9。长ncRNA传统上被定义为由至少200个碱基的RNA链组成10,11,12。这意味着识别了扩增子长度后,在设计这些靶标时,除了上述内容外,不需要特别考虑其它因素。
相比之下,microRNA(miRNA)家族成员则面临相当大的设计挑战。其为21-23nt的短非编码RNA(sncRNA),通过复杂的细胞途径在转录本处理的几个阶段产生。MicroRNA通过与转录本结合负面调节蛋白质翻译(见Kato等人在200813中的综述13),并诱导RNA诱导沉默复合物(RISC)形成14。商业化检测法,例如MystiCq®系列,是针对miRNA设计挑战的一个热门解决方案。已经有几种用于miRNA分析的qPCR方案15和无商业产品的生物学研究方案,有几篇文章描述了潜在的解决方案。其中许多依赖于通过连接一个衔接子(参见第22章,Casoldi等,PCR Technologies;Current Innovations en Nolan16)或通过polyA聚合酶(PAP)添加poly-A尾17,向原始miRNA添加碱基。在每个miRNA特异性引物中添加标签可以优化杂交Tm,且已经证明含有DNA引物的反应比加入锁定核酸的反应更有效18。在报告中,设计特异于miRNA的DNA引物,除了考虑常规引物指南,还需注意:
- 检查miRNA序列并忽略3’端的所有末端A碱基。• 将正向引物定为从5’端到3’端最后4个碱基的最长序列(忽略上面鉴定出的A碱基)。
- 正向引物的3’端的2个碱基之一优先选为A或T。
- 3’端的3个碱基优先选择包含1个或2个A或T。
- 3’端的5个碱基优先选择包含2-3个A或T。
- 使用最邻近算法分析正向引物的Tm。如果低于59°C,按给定顺序将下列碱基添加到5’端并计算每次加入后的Tm:G、A、C、G、C(产生CGCAGN18形式的引物,其中N是miRNA特异性碱基)。选择Tm最接近59°C的最短引物。如果温度高于59°C,则去除5’碱基并在每次去除碱基后计算Tm。选择Tm最接近59°C的最长引物。
- 选择反向引物的3’碱基,确保它们不与正向引物互补。按照正向引物的描述评估末端5个碱基。
- 向引物添加15×T(例如,5’ T15 N5 3’)。
- 使用最邻近算法分析正向引物的Tm。如果低于59°C,将下列碱基依次添加到引物的5’,并在每次添加后计算Tm:G、A、C、C、T、G、G、A、C、C(产生CAGGTCCAG T15 N5形式的引物,其中N是miRNA特异性碱基)。选择Tm最接近59°C的最短引物。
- RT引物是CAGCTCCAG T15 V N(其中V=A,C和G,N=A,C,G,T)。
引物设计实例
由于靶序列决定了引物序列,因此可能无法始终达到所需的设计标准。因此,需要通过实验特异性优化避免降低实验设计要求。在设计成功的实验前,一些PCR靶标可能需要进行特殊的处理。常见的案例涉及病原体的检测,包括病毒。众所周知,许多病毒基因组的特定位置具有高度的变异性。乙肝病毒是一个很好的例子。在最近一项研究中,为成功设计针对已知HBV变异19的qPCR实验,需要对所有可用的HBV基因组序列进行广泛的比对。使用ClustalW比对几百个序列,试图找到可用于通用实验设计的显著序列的共有序列。比对结果的一个片段如图6.4所示。星号(*)代表在所有基因组中分析中发现的共有核苷酸。(由于空间限制,未显示这项比对中大部分其它序列)。

图 6.4.HBV基因组数据的部分ClustalW分析。使用ClustalW比对所有已知的HBV基因组序列并鉴定出保守的核苷酸(*)。
在这种情况下设计引物和探针时,可能需要使用含有混合碱基的寡核苷酸,也称为“摇摆”或简并碱基。例如,考虑如图所示的HBV共有序列的细节(图6.5)。

图 6.5.HBV比对的选定区域显示共有区域
该区域引物大约需要二十三个碱基。在这种情况下,当考虑所有HBV基因组序列的所有可能性时,该二十三个碱基区域的实际序列如图6.6所示:
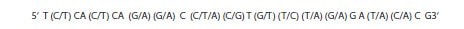
图 6.6.引物序列的排列需适应所选共有引物区域的所有碱基选项。
模糊碱基的位置可用混合碱基的标准单字母代码表示(表6.3)。当这些应用于图6.5和6.6所示的序列时,寡核苷酸可以如图6.7所示。
A =腺苷,C =胞苷,G =鸟苷,T =胸苷
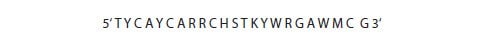
图 6.7.共有区域的寡核苷酸的模糊碱基由标准单字母代码表示。
该选择不太可能形成成功的PCR引物,因为使用大量简并碱基需要解决另外的考虑因素。尤其是使用该序列制备的合成寡核苷酸,实际上会产生每种可能的单碱基序列的混合物。可能的单个引物序列的数量通过乘以每个位置的各个碱基数来计算。对于该序列,这意味着1×2×1×1×2×1×1×2× 2×1×3×2×1×2×2×2×2×1×1×2×2×1×1 = 6,144个可能的寡核苷酸。因此,反应中每种特定寡聚物的有效浓度按比例减少。经验分析表明,引物中不同序列排列的数量不应超过512,因此这个例子不是最佳的简并碱基引物。在不同的位置重新设计引物是一个可能的解决方案。在如图6.8所示的例子中,引物包含2个潜在的错配碱基。但是,它们各自有一个替代碱基,在混合物中产生4个寡聚核苷酸,且简并位于引物的5’端区域。这些因素提供了比图6.7所示的引物更高的成功几率。

图 6.8.在共有区域比对共有碱基和潜在引物位置的序列该共有引物使用简并代码显示。
当在PCR中使用简并寡核苷酸时,可能需要改进的扩增方案。循环可以从在低退火温度(35-45℃)下进行2-5个循环开始。此外,从退火温度到延伸温度缓慢上升坡道需具体化,大约需要3-5分钟才能达到延伸温度。然后应在更严格的退火温度下以25-40个循环完成方案,而不进行坡道修饰。
在可能的情况下,尽量避免核苷酸异质性。如果无法避免异质性区域,通常是在有困难靶标情况下,则使专门的寡核苷酸修饰,例如肌苷和其它“通用”碱基,例如5-硝基吲哚,可能有助于降低复杂性;添加修饰基团,如锁定核酸(参见定量PCR和数字PCR检测方法)可能提高性能。
引物修饰
可以将修饰的核苷酸掺入到PCR引物中。加入一个锁定核苷酸是常见实例。锁定的核苷酸是一个修饰RNA核苷酸。锁定核酸的核糖部分用一个额外的桥连接2’的氧和4’的碳进行修饰(参见定量PCR和数字PCR检测方法)。这种桥将核糖“锁定”在3’内切构象中。必要时,锁定的核酸可以和寡核苷酸中的DNA或RNA碱基混合。锁定核酸的修饰使得热稳定性增加,允许设计Tm较长的未修饰的引物相相等的更短探针。含有锁定核酸的序列比仅有DNA组成的寡核苷酸特异性更强,非常适合SNP检测。锁定核酸修饰的其他应用包括为复杂序列设计寡核苷酸,例如因具有高度变异性而难以设计通用检测方法的病毒22。
探针设计
关于PCR引物,qPCR探针设计也在很大程度上取决于序列内容和所需的应用。单探针,如双标记探针和分子信标,通常长度为20-30个碱基。Scorpions®探针长度较短,为15-25个碱基。在LightCycle或FRET系统中,有两个探针位于紧邻位置,由1-5个碱基隔开,分别为传感探针(探针1)和锚定探针(探针2)。
- 当使用双标记探针时,Tm应比引物高7°C至10°C,以确保探针在引物杂交和延伸之前与靶标结合。反应中的FRET探针也是如此。用于SNP检测时,传感探针(探针1)位于错配位点上方,避开探针末端的3个碱基,并且Tm值比锚定探针(探针2)低(约5°C)。Scorpions®探针是该建议的例外情形,因为探针与延伸后的新合成的模板结合(而不是和之前的结合,就像其他探针系统一样)。
- 对于使用双标记探针的定量研究,目标是将探针定位在靠近正向引物3’端但不重叠(约5个碱基)的位置;对于SNP检测,将双标记探针或分子信标置于扩增子的中心,并将SNP置于探针的中心。
- 避免在探针的5’端,靠近报告基因处使用胍,因为这会导致淬灭。
- 确保探针序列中G少于C。
- 避免运行相同的碱基(<4)和回文序列。
- 确保探针不会形成二级结构。
- 确保探针不能与引物杂交。
- 当设计用于多重反应的探针时,确保所有的探针和引物之间没有潜在的相互作用。
分子信标
在选择了合适的探针区域后,将互补的茎添加到探针的5’和3’末端以产生分子信标结构20。以下例子显示了将茎序列(红色)添加到双标记探针中以产生分子信标(图6.9摘自Thelwell 200021)。

图 6.9.将双标记探针修改成分子信标形式牛津大学出版社的核酸研究。经牛津大学出版社版版权许可中心许可,可以书籍/电子书的形式重复使用。
Scorpions® 探针
Scorpions®探针需要将探针与正向引物组合,使得它们采用以下结构:5’染料茎-探针-茎-淬灭剂-阻断剂-引物。引物和探针必须位于相反的链上,因为探针与新产生的模板结合,该模板与引物位于同一条链上。如图6.10所示,将标记,淬灭剂,PCR阻断剂和茎序列添加到双标记探针来创建Scorpions®探针摘自Thelwell 200021)。

图 6.10.将双标记探针测定修改为Scorpions®探针形式。牛津大学出版社的核酸研究。经牛津大学出版社版版权许可中心许可,可以书籍/电子书的形式重复使用。
探针修饰
当探针位于含有异常异质性的序列区域时,如PCR引物中所述管控模糊碱基。类似地,可将锁定核酸添加到探针中,其原因与在引物中添加一样。
双标记探针、分子信标或Scorpions®探针的5’端标记有荧光团,单次分析通常用6-FAM™,多重分析通常按FAM、HEX™/ JOE™、青蓝素5的顺序选择使用(确定标记与仪器的兼容性至关重要)。双标记探针或分子信标的3’端和Scorpions®探针内部茎区的3’末端用淬灭基团分子修饰。以往,染料TAMRA用于FAM发光的受体,导致FAM淬灭。暗淬灭剂技术的发展使得Black Hole Quenchers™和我们最近推出的Onyx Quencher™系列广泛采用(参见定量PCR和数字PCR检测方法)。
模板对照
使用相对较短的扩增子(通常短于150个碱基)的一个优点是,可以进一步合成长寡核苷酸,然后作为合成扩增子的靶标。使用这样的靶标有助于开发和优化预期靶标可能很稀少或不足的实验,例如抑制对照实验SPUD23(见附录A)或传染性疾病研究19。
寡核苷酸的合成和处理
订购用于PCR的定制寡核苷酸时,须根据所需合成产率/规模、纯度和所需的修饰判定产品。这些因素会相互影响,例如,纯化水平越高,寡核苷酸的质量就越好,代价则是会降低总产量。表6.4,6.5和6.6提供了我们关于生产寡核苷酸的合成规模和预期产量的指导。
寡核苷酸纯化
当合成DNA时,每个核苷酸从序列的3’末端开始依次与生长链偶联。在每个偶联循环中,少量寡核苷酸链不会延伸,结果形成全长产物和截短序列的混合物。当寡核苷酸从载体上被切掉,保护基团被移除后,通过纯化将全长产物从截短的序列中分离出来。通常,特定应用所需的纯度取决于存在的截短低聚物可能的影响。对于某些应用,仅存在全长(n)寡核苷酸十分关键的。对于PCR引物等其他应用,存在较短的寡核苷酸(n-1,n-2,...)可能不会影响实验结果。
脱盐纯化
脱盐程序可除去合成、裂解和脱保护步骤的残余副产物。
对于包括PCR在内的许多应用,对长度超过35个碱基的寡核苷酸脱盐纯化是可接受的,因为寡核苷酸占绝大多数,超过了较短产物的其他任何影响。克隆所需的寡核苷酸或长度超过35个碱基的需要另一种纯化方法,例如反相滤芯纯化(RP1)、HPLC或PAGE(取决于长度)。
反相柱纯化(RP1)
反相柱上的分离去除了大部分截短的序列。这种分离方法基于全长产物和截短序列之间的疏水性差异。全长寡核苷酸保留在柱子上时,而截短的序列被洗掉了。然后,将所需的全长产物洗脱下来,并从柱中取出。
反相HPLC
随着寡核苷酸的长度增加,截短序列的比例也趋于增加。并非所有这些杂质都会被RP1去除,因此对于较长的寡核苷酸,例如人工扩增子模板寡核苷酸或标记的探针寡核苷酸,建议使用HPLC或PAGE纯化。反相高效液相色谱(RP-HPLC)的操作原理与反相色谱柱相同。但是,更高的分离度可实现更高的纯度水平。HPLC是一种有效的纯化荧光(如qPCR探针)标记寡核苷酸的纯化方法,因为它们固有的亲脂性能很好地分离产品与污染物。此外,由于色谱柱的容量和溶解性质,RP-HPLC的使用范围较广。基于亲脂性的分离度会随着寡核苷酸的长度而降低。因此,通常不建议RP-HPLC用于纯化超过50个碱基的产品。虽然较长的寡核苷酸(最多80个碱基)可以使用这种方法纯化,但可能会对纯度和产率有不利影响。
阴离子交换HPLC
阴离子交换分离基于分子中磷酸基团的数量。阴离子交换纯化方法是在季铵固定相柱或类似结构上使用盐梯度洗脱。该分辨率非常适合于量少的纯化。这项技术可以和RP-HPLC联合使用,为分离过程增加第二个维度。阴离子交换HPLC受寡核苷酸长度限制(通常最高为40mer)。寡核苷酸越长,在阴离子交换HPLC柱上的分辨率就越低,因而靶标寡核苷酸的纯度就越低。
聚丙烯酰胺凝胶电泳(PAGE)
PAGE分离基于电荷而非分子量,因而有良好的尺寸分辨率,可产生纯度水平为95-99%的全长产物。从凝胶中提取寡核苷酸和除去绝大多数截短的产物所需要的程序复杂,所以PAGE的产率低于其他方法。当需要高纯度产品时,推荐使用该方法。纯化较长寡核苷酸(≥50碱基)时,推荐使用PAGE。
*确保20mer以上长度。较短的寡核苷酸可能OD值更小。
*确保20mer以上长度。较短的寡核苷酸可能OD值更小。
注意:合成后修饰可能比上述值少50%。
所有探针都是通过RP-HPLC纯化。
寡核苷酸制备
提供干燥的DNA寡核苷酸,即可通过重悬浮后使用。建议将寡核苷酸重悬浮于弱缓冲液中,比如TE(10mM Tris,pH 7.5-8.0,1 mM EDTA)。在不适合使用TE的应用中,可使用无菌无核酸酶的水。但是,高级别的水可能是弱酸性的,不建议用于长期储存寡核苷酸。
通过以下指南可获得100μM原液:取所提供的摩尔数(nmol)(见在寡核苷酸随附的试管标签和/或质量保证文件)并乘以10。按照该结果添加相应微升的液体到试管中,使最终浓度为100μM。例如,如果寡核苷酸产量为43.5nmol,则添加435μL体积形成100μM原液。请注意,这相当于100pmol/μL的原液。该原液可以根据需要进一步稀释,依据具体应用的需求。对于PCR,通常使用10μM或20μM的工作浓度。将原液在-20°C下分份存储,避免多次冻融循环。
参考文献
如要继续阅读,请登录或创建帐户。
暂无帐户?