聚合酶链反应:
聚合酶链反应(PCR)1,2,3已成为分子生物学应用最普遍的技术之一。用于从基础研究到高通量筛选的应用。它是一种强大的技术,但与此同时,它简单易用,成本较低,因此其应用普遍而多样。该技术用于从少量DNA或RNA源扩增特异性靶DNA片段(在逆转录步骤后产生互补DNA(cDNA),请参见逆转录反应)。PCR的一个主要优点是靶序列可以从起始材料一个副体开始扩增,甚至当模板被降解并被抑制剂污染时也能如此。例如,已对从古埃及木乃伊中提取的DNA进行了研究,以确定图坦卡蒙国王家族的成员4。大型电影和大多数犯罪系列电视剧中都在“调查”的某个阶段有关于PCR的内容,尽管有些过于虚构。
PCR循环过程
典型的PCR包括:
- 初始变性:将反应温度升至95 °C,孵育反应物2-5分钟(根据酶特性和模板复杂性,最多10分钟),以确保所有复杂的双链DNA(dsDNA)分子被分割成单链以便扩增。
- 循环:
- 变性:将反应温度升高至95 ℃,其将所有dsDNA解链(破坏互补碱基之间的氢键)成单链DNA(ssDNA)。
- 退火:将温度降低至低于引物的解链温度(Tm)约5 ℃(通常为45-60 ℃),以促进引物与模板的结合。
- 延伸:温度升至72 ℃,这是DNA聚合酶活性的最佳温度,在这个温度杂交的引物被延伸。
- 变性:将反应温度升高至95 ℃,其将所有dsDNA解链(破坏互补碱基之间的氢键)成单链DNA(ssDNA)。
- 重复:步骤1-3循环进行,使扩增子以指数扩增(图2.1和2.2)。
PCR由反应物加热和冷却循环组成。每个温度平台用来控制反应的确定阶段,孵育时间取决于仪器、反应板或管以及试剂。这些应针对每个实验进行优化,特别是在需要提高检测灵敏度的情况下5。
初始变性阶段包括高温期,在此期间复杂双链DNA(dsDNA)的二级结构被解链成单链DNA(ssDNA)。DNA在这个阶段经受高温,该阶段需要足够长以分离所有链,从而它们可用于引发,但又不能长到DNA被损坏。初始(或后续)变性步骤中的降解或不完全变性导致检测灵敏度下降5。
在较短的变性步骤中,启动循环(在95℃下10秒至1分钟),靶序列的DNA链分离形成单链,正如在初始变性阶段一样。然后将反应物冷却至引物退火温度。
退火步骤(30秒至1分钟,温度45-60℃)是必须的,以使引物与每条DNA单链上的互补序列结合。引物的设计应使得它们包围感兴趣的靶标,位于它们之间的序列区域称为扩增子。通常,退火温度可以估计为比引物-模板DNA双链体的解链温度低5℃。
最后阶段是延伸步骤(在72℃下20秒至1分钟),该步骤使得DNA聚合酶将引物序列从每个引物的3'延伸至扩增子的末端。1分钟的延伸通常足以合成多达2千碱基(kb)的PCR片段。为了扩增更大的片段,可以按每kb 1分钟的比例延长延伸步骤。在第一次延伸期间,模板不限制长度,因此模板的合成会超过扩增子长度。在随后的延伸步骤中,扩增子长度将由每端的引物序列限定。
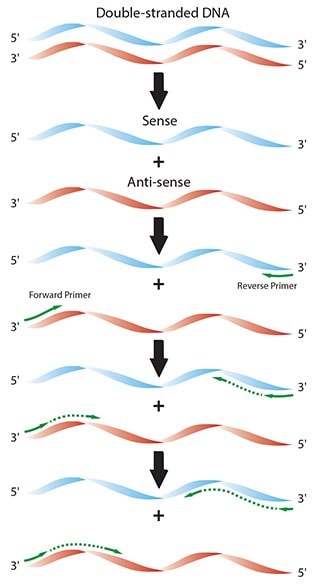
图 2.1.典型PCR中各个反应过程的示意图

图 2.2.在理论PCR中,靶扩增子的量随每个循环加倍,导致靶序列以指数扩增(X轴显示PCR循环,Y轴显示扩增子分子的总数)。
在第二个循环开始时,有两种形式的模板在反应中:原始DNA链和新合成的DNA链,由引物序列组成,随后是在3'末端延伸的可变长度的扩增子。在第一个循环中未引发的剩余原始DNA链可以在第二个循环中被捕获,并产生由引物和延伸产物组成的分子。来自第一个循环的引发和延伸的分子将是与新合成材料互补的引物的模板。
在第三个循环中,由第二个循环产生的新合成的靶区域DNA仅包含扩增子,因此成为特异性模板。
连续重复循环,使复制序列以指数扩增(图2.2)。所需的循环数取决于PCR产物的所需产率。这又取决于初始起始拷贝数和扩增效率。由于起始材料与最终产物的相对浓度非常低(例如,如果单个分子经历40个扩增循环并且在每个循环达到100%效率,则为1/5.511 ,图2.1),在反应结束时得到的DNA几乎完全是PCR扩增子。
虽然理论PCR应产生连续指数扩增,然而反应最终会达到平台期。这似乎是由于DNA聚合酶与DNA产物的非特异性结合6,7。当使用定量实时PCR(qPCR)和监测荧光标记的变化时(请参见定量PCR),这清楚可见。
在反应结束时,使用凝胶电泳分析扩增产物。根据产生的量和扩增片段的大小,可以通过用溴化乙锭染色凝胶(图2.3)或使用最近推出的染料(例如BlueView™)直接观察反应产物8。
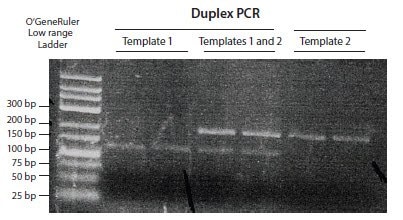
图 2.3.通过用溴化乙锭染色的琼脂糖凝胶解析的常规PCR产物的实例。进行了双重PCR以同时检测2个靶,每个靶的大小不同。两个片段都可以看作是不同的条带(图片由MarionGrieβl友情提供)。
PCR测定的组成
以下是PCR所需的部件摘要。其中许多都在专著中有更加详细的描述,亦可在本指南末尾详细的标准PCR实验方案(附录A)中看到。
模板
有几种可用于纯化RNA和DNA的方法。虽然这些方法都合适,但它们可能不尽相同。如果模板受到来自样品源的其他材料的污染,可导致PCR抑制,在极端情况下,可使测定完全失败。有一些实例证明,污染物甚至可能导致反应增强9,10。有关模板纯化和质量控制的更多信息,请参见样品纯化和质量评估;有关逆转录(RT)反应的更多信息,请参见逆转录。通常,将10 ng至1 μg的基因组DNA(gDNA)添加至20-100 μL PCR测定中,而将cDNA合成反应物稀释1/5至1/10,并将5 μL添加至20-50 μL反应物中。
引物
寡核苷酸引物的长度通常为15-25个核苷酸,具有40-60% GC,每
对成员都有相似的Tm。评估短寡核苷酸(<25个碱基)的Tm的简单方法是使用公式11:
Tm = 4(G + C) + 2(A + T)
关于底物设计和处理的更多信息,请参见PCR/qPCR/dPCR测定设计。用于退火的理想起始温度估计为比解链温度低5℃。可以使用温度梯度PCR仪优化退火温度。附录A给出了温度优化的实验方案(以qPCR为例)。
DNA聚合酶
存在多种DNA聚合酶,根据实验定义选择正确的酶(而不仅仅是实验室冰箱里可用的酶)是至关重要的。例如,一些酶被设计为在低温下无活性,旨在用于热启动实验方案,以减少非特异性扩增,而有些酶则具有低错误率,选择用于扩增片段进行克隆。
dNTP
PCR中每种dNTP(dATP、dCTP、dGTP、dTTP)的浓度通常为200 μM。然而,dNTP浓度必须过量使用,而且对于长片段或高丰度靶标的扩增,还可能需要增加更多用量。最近,对PCR缓冲液组分进行了改进,例如CleanAmp™ dNTPs12。这些是经过修饰的核苷三磷酸,其阻断DNA聚合酶核苷酸掺入。 CleanAmp dNTPs通过初始加热步骤和随后的典型热启动循环条件下的变性步骤激活。该过程限制了每个PCR循环期间可用的活化dNTP的量,这使得更特异和更有效地扩增所需产物。反过来,它们减少或者完全避免了错误引发或引物二聚体形成,因此是热启动DNA聚合酶的替代方案。
MgCl2浓度
需要游离的Mg2+离子作为DNA聚合酶活性的辅助因子;然而,Mg2+离子与dNTPs、引物和DNA模板形成复合物。因此,需要通过测试含有不同浓度的反应物来为每个实验选择最佳的MgCl2浓度。MgCl2浓度通常在1.5-5.5 mM之间,但可以在优化期间(请参见测定优化和验证)在1-8 mM范围内测试。
荧光标记
当要直接检测扩增子时,例如当使用qPCR或数字PCR(dPCR)设置时,荧光染料被掺入反应物中。它们包含在缓冲液中或附着在引物或其他探针上(请参见定量PCR和数字PCR检测方法)。
仪器
最初,进行PCR是劳动密集型的工作,因为它是通过在3个水浴之间物理移动反应管来进行的,每个水浴设定在变性、退火和延伸所需的温度。将油加入到反应物的顶部以防止蒸发,还要在不受油干扰的情况下进行分析,是件棘手的工作。如今,通常使用市售的许多专用热循环仪器进行。板和仪器的形式多选,有48、96或384孔,并且与多通道移液器兼容。大多数人使用加热的盖子,无需油盖。然而,有基于原始水浴原理的高通量仪器13。
特定应用专用PCR
有各种专用PCR需要对反应物进行相应修饰。这里将介绍一些针对流行应用所做的修饰。
克隆
PCR可用于扩增所选序列以插入载体中。可以对这些序列加以修饰,以包括用于克隆酶识别的特定区域(尾部)。针对载体的引物用于分离已经克隆到载体中的片段。具有低错误率的DNA聚合酶是PCR步骤所必需的。
序列标记位点
它们被设计到引物的5'末端,在高通量测序期间使用。在高通量测序中,需要特定标记作为特定样品基因组的特定区段存在于所选克隆中的指示物,使得基因组序列数据可以被去卷积,关联于原始源样本。
定点诱变
为了研究蛋白质功能,可以将期望的突变引入DNA序列中。将所需的碱基变化设计到含有所需的克隆酶限制性位点的引物中(朝向5'末端)。另一种方法采用多步骤实验方案,其中对靶标特异并含有克隆位点的引物与含有所需突变的引物一起使用。
产品列表
参考文献
如要继续阅读,请登录或创建帐户。
暂无帐户?