定量PCR基础知识
定量PCR(正式名称为实时定量PCR,qPCR)检测在标准PCR技术基础上演变而成,可用于计算样本的起始材料量。由于在反应进行时即可检测产物,qPCR相比常规的终点PCR具有更大的检测动态范围;一次运行可检测单个至大约1011个拷贝。实时定量PCR和后续的扩增子检测都在封闭的试管中进行,无需凝胶电泳等PCR后操作,极大降低了交叉污染风险。
本页将介绍qPCR基本原理。常用检测化学原理参见“定量PCR和数字PCR检测方法”。
基础研究
qPCR使用荧光报告染料间接检测每次扩增循环中的核酸量。荧光信号的增加与反应(聚合酶链式反应)重复阶段中产生的指数累积的PCR产物(扩增子)量成正比(参见聚合酶链式反应)。报告染料分为:双链DNA(dsDNA)结合染料,引物偶联染料,或其他染料偶联寡核苷酸,即探针(参见定量PCR和数字PCR检测方法)。
dsDNA结合染料如SYBR® Green I代表了最简单的检测化学形式。当在溶液中游离或仅存在单链DNA(ssDNA)时,SYBR Green I染料所发光信号强度较低。随着PCR进行,dsDNA增加,更多染料结合到扩增子上,因而信号强度增加。
另外一种是探针(或根据检测化学使用两者的组合),由于它结合在引物之间的模板特定区域,相比dsDNA结合染料它的检测特异性水平更高。最常用的探针是双标记探针(DLP,又名水解或TaqMan®探针)。DLP是一种寡核苷酸,5’端标记荧光,如6-FAM™ ,3’端标记淬灭分子,如暗淬灭剂BHQ®1或OQ™ (参见定量PCR和数字PCR检测方法)。这类探针经过设计可与两种引物之间的模板杂交,并与固有5'至3'外切核酸酶活性的DNA聚合酶结合使用。DLP在溶液中游离时,因为报告染料非常接近淬灭剂基团,信号强度较低。随着反应产生更多模板,更多探针与模板杂交,而模板又被前进的DNA聚合酶利用5'至3'外切核酸酶活性切断。荧光信号强度随着5’端报告染料在溶液中的释放而提高。使用不同报告染料标记的探针可在一次(多重)反应中同时检测和定量多个靶点。
典型的qPCR运行由交变温度孵育的重复循环组成,参见循环程序3.1。一般当qPCR选用dsDNA结合染料、分子信标或Scorpion®探针的化学检测时会使用这种程序。引物延伸在72°C时最高效,因为这是大多数DNA聚合酶持续合成的最佳温度。72°C的聚合速率约100个碱基/秒。但较低温度下的持续合成力足以扩增较短模板。qPCR扩增子(<200碱基)通常短于常规PCR产物,因而使用双标记探针时,延伸和退火一般在60°C下结合为一步进行(循环程序3.2)。
循环程序3.1
标准qPCR程序示例,使用dsDNA结合染料、分子信标或Scorpions®探针检测。
- 初始变性:反应温度升高到95°C,样本孵育2-10分钟(时间取决于聚合酶热启动机制),确保所有复杂靶点(dsDNA)都分离为可扩增的单链。
- 循环:
- 变性:反应温度升高到95°C,持续10秒以熔解全部dsDNA。
- 退火:温度降低到60°C,持续30秒以促进引物和探针(如有)与模板结合。
- 延伸:随后延伸温度升高到TaqDNA聚合酶持续合成力最高的72 °C。延伸持续时间取决于扩增子大小(30秒/1kb)。延伸时间取决于所需扩增子长度及所用酶。由于qPCR扩增子较短,时间通常为5-30秒。
- 变性:反应温度升高到95°C,持续10秒以熔解全部dsDNA。
- 重复:步骤1-3重复,通常循环40次。
循环程序3.2
DLP检测的两步循环qPCR程序示例。
- 初始变性:温度升高到95°C,孵育2-10分钟(取决于聚合酶的热启动性)。
- 循环:
- 变性:反应温度升高到95°C,持续10秒以熔解全部dsDNA。
- 退火和延伸:温度降低到60°C持续30秒以促进引物与模板结合。由于此温度下的DNA聚合酶活性充分,后续延伸自然发生。
- 重复:步骤1-2重复,通常循环40次。
反应过程中的荧光变化通过实时定量PCR热循环仪测量。专用的实时PCR仪将温度循环与光学元件结合。光学元件提供合适波长的光激发荧光团并检测生成的发射光。许多现代化仪器都设有固定的显示屏或在附近安装电脑显示器,方便追踪反应进程(图3.1)。
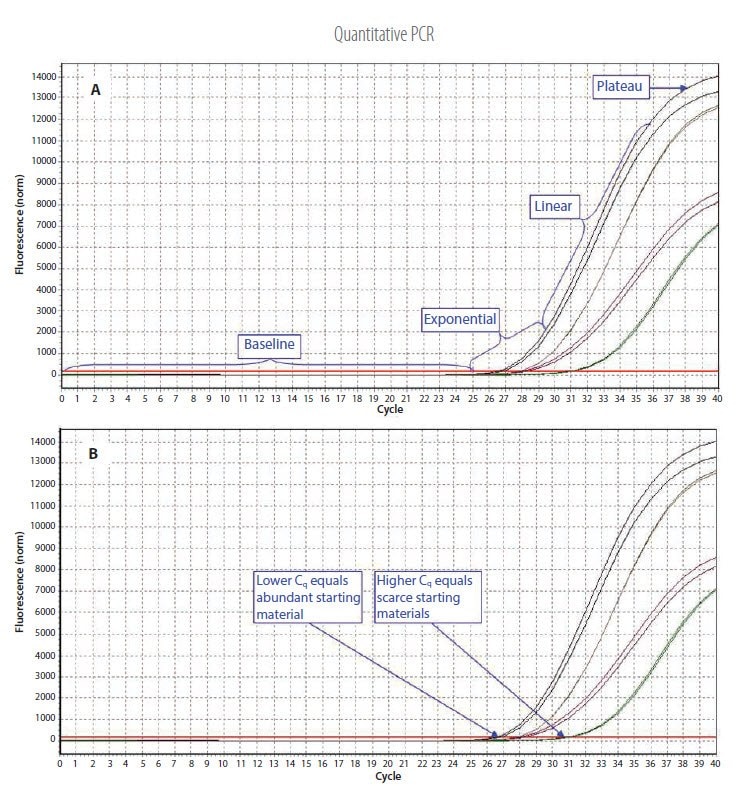
图 3.1.qPCR分析数据示例。A)反应的不同阶段。基线:模板初始浓度低,因此荧光强度太低而不能被检测到,只有背景信号明显存在。指数:在靶样产量达到检测阈值(显示为红色阈值线)后,可在整个指数阶段可以跟踪反应过程。线性:随着模板浓度增加,可用DNA聚合酶浓度减少,反应速率降低。平台:反应处于最大产量期。B)个体反应的表征使用荧光首次超过阈值时的循环数——定量循环(Cq)。若起始材料丰富,会在较早循环观察到扩增且Cq值偏低。如起始材料稀缺,会在后续循环观察到扩增且Cq值偏高。荧光、Cq和扩增产物量之间的这种相关性使得模板定量的动态范围更大。
gDNA靶点的定量和分析
实时荧光定量PCR十分适合gDNA靶点分析。包括基因分型/SNP
检测、甲基化分析、筛选转基因序列或监测插入和缺失。
mRNA转录本的定量和分析
qPCR的一个常见应用是基因表达分析,例如比较对照和处理样本之间目的基因的mRNA浓度。mRNA可通过两步法或一步法RT-qPCR进行定量(参见反转录实时定量PCR了解RT反应细节)。
两步法RT-qPCR
反转录和qPCR反应依次在不同试管中进行。以总RNA或较少使用的
多聚(A)+RNA作为起始材料,使用反转录酶延伸oligo-dT引物、
随机引物、oligo-dT引物/随机引物混合物或基因特异性引物合成cDNA。反应物等分用于qPCR反应。
一步法RT-qPCR
反转录和qPCR反应在同一试管进行使得实验设置简化
,且由于样本无需后续处理,污染风险也随之降低。
ncRNA转录本的定量和分析
大多数非编码RNA都超过100个核苷酸,因此可利用与mRNA分析相同的技术进行RT-qPCR
检测和定量。比较而言,较短的非编码RNA比如micro和piwi RNA基本上只有单个PCR引物那么大。因此在进行qPCR之前,需使用技术手段延长这些短RNA。商品化系统采用了两种主要方法:1)使用茎环RT引物和2)多聚腺苷化后再加入oligo-dT接头引物进行反转录(图3.2)。Casoldi等人开发了另外一种没有商品化的方法,使用接头分子与样本中所有RNA连接,并用特异引物区分各靶点1,2。
茎环RT引物法由Applied Biosystems(目前属于Life Technologies/Thermo)公司开发并商品化,用于后续的TaqMan探针法qPCR3。如图3.2A所示,RT引物的5’端可与距离自身3’端几个核苷酸位置的碱基配对,形成由非配对环分隔的碱基配对茎。除了3’端最后几个核苷酸,RT引物的所有核苷酸都是通用的,即所有miRNA引物中都含有相同序列。茎3’端伸出的最后几个核苷酸与目标miRNA的3’端互补。引物沿着miRNA模板延伸合成
cDNA,再由miRNA特异性上游引物和通用下游引物进行扩增,后者与茎环RT引物的5’端
互补。
其他所有miRNA qPCR方法,包括我们的MystiCq®品牌,如Shi和Chiang4所述都通过多聚腺苷化加长miRNA。多聚腺苷酸聚合酶(PAP)是一种不依赖模板的酶,可催化腺苷残基从ATP转移到任意RNA的3’端。接着可使用oligo-dT引物进行反转录,如图3.2B所示。oligo-dT引物的5’端含有接头序列,借助与特异miRNA互补的上游引物和与接头序列互补的下游引物可进行后续的qPCR。
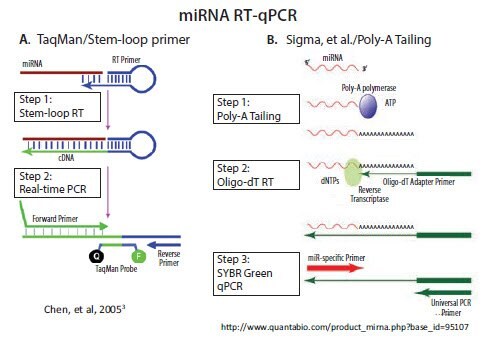
图 3.2.miRNA的RT和qPCR有两种商品化方法。A)环状引物与miRNA的3' 末端杂交后引发RT步骤。通过环进行的PCR延伸形成了组合的miRNA和通用序列,长度足够用于扩增 B)miRNA加上的多聚腺苷酸尾为由oligo-dT和通用引发序列组成的引物提供了引发位点。来自牛津大学出版社《核酸研究》。经由美国版权结算中心获得牛津大学出版社以书/电子书形式重新使用的许可后转载。
两种商业方法各有优缺点。茎环引发适用于两步法RT-qPCR,而大多数多聚腺苷化法需要额外的RT步骤才能进行qPCR。某些产品可将多聚腺苷化和RT结合在一次反应中,用于进行两步法PAP加尾/RT-qPCR。但这种方法的检测灵敏度可能会减弱(未发表内部结果)。相反地,多聚腺苷化后RT可生成稳定的cDNA池,可立即或在以后任意时间点用于检测所有microRNA、mRNA或其他RNA。使用茎环/TaqMan法,每种miRNA检测都需要不同的引物。同一RT反应可加入多种茎环引物5,多种miRNA进行多重RT反应的RT引物池可从Life Technologies购买。然而,两种方法都不能用于较晚发现的miRNA的qPCR检测,也不能用于来自相同cDNA的mRNA的qPCR检测。Castoldi等人所述系统1,2的独特性在于它确实提供了针对同一总RNA样本中mRNA、前体和完全加工miRNA分子的检测方法。
qPCR要求
仪器
许多qPCR仪器都经过特别设计用于支持特定范围的应用,比如ABi 7900高通量PCR仪使用自动上样的384孔板,而Illumina生产和销售的Eco仪则支持单个48孔板。因此要选择满足研究需要的最适用仪器。仪器装载软件最好选择用户友好型,这种软件运行最理想的功能并且数据输出形式灵活,方便下游统计分析软件包轻松处理数据。如此人员培训时间减少,因而能更快获得结果。其他需要的配置包括绝对均一的PCR模块(96个复孔的绝对最大偏差为1Cq=2倍)以及宽光谱下极致灵敏、均衡激发和检测发射光的光学系统。如此便于选择更多的荧光团和进行多重反应。还要考虑特定耗材相关的运作成本,比如反应使用的不是标准微孔板,就要考虑非标准上样板子或管子的便利性。
模板
qPCR起始仅需极少量的目标核酸拷贝(约等于100pg gDNA或cDNA)。为了尽量减少反应抑制剂的污染,起始模板量应保持在实现准确定量所需最低用量。起始材料是RNA时,引物设计和DNase I处理可能会引入gDNA污染而导致信号减弱。
引物
在qPCR中,无论使用dsDNA结合染料还是基于探针的检测化学,设计高质量的引物都是最重要的实验前步骤之一。有关检测设计的详细讨论参见PCR、qPCR、dPCR检测设计。为了使灵敏度和特异性达到最高,引物应包含任何所要求的修饰,例如锁核酸(参见qPCR检测化学)。
探针
使用探针法进行扩增子检测时,引物二聚体和非特异性产物虽然不会检出但应尽量避免,因为它们会降低反应效率。为了使灵敏度和特异性达到最高,应选择适合应用的探针类型并包含任何需要的修饰,例如锁核酸(参见qPCR检测化学)。
dNTP
标准PCR和qPCR预混液包含dATP、dCTP、dGTP和dTTP。然而,有些可用的混合物用dUTP代替了dTTP。使用dUTP进行的先前反应的产物将含有尿嘧啶而不是胸腺嘧啶。它们易被尿嘧啶DNA糖基化酶切断(UNG)。因此后续反应加入UNG预先孵育可防止反应之间的携带污染。为了效率所有实验室进行的PCR都应使用dUTP。
镁
氯化镁(MgCl2)对于逆转录酶、Taq DNA聚合酶和Taq DNA 5’至3’外切酶活性都是必需的。含DLP反应的最佳Mg2+浓度通常是3–6mM。大部分预混液都含有MgCl2,但有时需要对浓度进行优化,因此预混液产品通常含有额外一管纯MgCl2(参见检测优化和验证)。某些情况下可能需要不含MgCl2的反应混合物,从而可以使用低浓度,例如使用Scorpions®探针检测时。
反转录酶
提供高产量cDNA、同时在高温下保持活性的逆转录酶对RT-qPCR的成功至关重要。酶的高温特性可确保RNA重要二级结构区域去稳定并易于杂交和后续扩增。进行一步法RT qPCR时,高温特性可搭配高解链温度(Tm)的基因特异性引物,增加反应特异性。两步方案重要的是确保酶从RNA获得线性成比的cDNA产量(RT评估详情参见逆转录)。
Taq DNA聚合酶
与为RT选择最合适的反转录酶一样,选择合适的聚合酶也十分重要。天然Taq DNA聚合酶的根本问题是低温下仍留有活性。这种残留的聚合酶活性导致非特异性引物结合形成非特异性产物。抗体阻断或化学阻断的Taq DNA聚合酶(“热启动”)通过阻断高温变性步骤前的酶活性矫正这一情况。
缓冲液
缓冲液或反应预混液通常含有dNTP、Taq DNA聚合酶、MgCl2和稳定剂。根据检测化学、仪器和反应要求,也可能含有SYBR Green I染料、ROX™、荧光素和惰性上样染料(参见上样控制染料)。PCR缓冲液组分和稳定剂通常是制造商专有的。分别购买可实现最大的灵活性,因为每种成分都可以在反应中单独优化。比较而言,虽然购买全部成分混合的预混液降低了灵活性,批次一致性却提高了,而且减少的移液步骤提高了便利性又降低了错误和污染的可能性。
上样控制染料
一些实时PCR循环仪需要在每次反应中加入ROX之类的上样染料来控制光学系统的变异并归一化信号强度差异。同样地,一些热循环仪进行SYBR Green I染料检测时(背景很低),需要初始荧光素信号构建虚拟背景。这些染料可在预混液中提供,也有可用于配制恰当浓度的单独组分提供。
MIQE指南
qPCR的一个明显问题是它极易产生易解读为定量结果的数字。这一点同样有误导性,因此可能需要质量控制和深入分析区分由不恰当检测设计、执行或数据分析造成的可能杂带。在没有充分的质量控制下发表显然是很重要的数据会导致报告不具有生物学或临床相关性,这还是最好的情况,最坏的情况是报告结果根本不准确。
从样本采集到qPCR数据分析,qPCR实验过程中的每一步骤都易受变异性影响6。因此样本选择和处理、核酸提取(会导致质量差异使得qPCR靶点检测缺乏重现性),检测设计和优化(会导致检测效率和灵敏度差异)以及数据分析(需要主观解释并使用适当的统计法)期间都可能会发生变异。关键的是提供给科学受众的信息要包含充分信息以便对实验进行严格评估7,但显然情况并非总是如此8。“发表实时定量PCR实验所需提供的基本信息”(MIQE)指南9解决了这些问题。MIQE指南旨在增加定量PCR实验的透明度,通过规定检测所需最基本信息让读者可以评估出版物上技术的有效性,并为新的qPCR检测的设计、优化和验证提供指导。
MIQE分为9个部分(实验设计、样本、核酸、逆转录、靶点、引物和探针、检测细节、PCR循环和数据分析;访问我们的MIQE页面下载核查清单,表1)。所有参数都与实验设计、优化和验证过程应收集的信息有关。使用商品化或出版物来源的检测时,仍应进行优化和验证。MIQE含有一张核查清单,其中标为必需(Essential)的要素对于充分描述qPCR检测是必不可少的,标为建议(Desirable)则表示这些信息有用,但就算没有也能复制检测方法及评价出版物数据。重要的是要认识到报告每次检测的PCR效率、分析灵敏度和特异性是必需的,并且应由研究者使用实验室条件加以确定。进行数字PCR(参见数字PCR)有特定的考虑因素,最近一份数字PCR MIQE出版物有列明10。
您符合MIQE要求吗?
发表实时定量PCR实验所需提供的基本信息
aClinical chemical Copyright 2009 美国临床化学协会。经由版权结算中心获得美国临床化学协会的网络发布许可后转载。
b所有必需信息都要跟稿件一起提交,建议信息如有也应提交。若引物来自RTPrimerDB数据库,从库中可获得qPCR靶点、寡核苷酸、实验方案和验证信息。
c首次提取RNA时进行没有反转录的检测评估DNA无残留十分重要。样本验证过不含DNA后,建议设立无反转录质控品,但不再是必须的。
d强烈建议公开探针序列。但由于不是所有商品化预设计检测的供应商都提供了这一信息,它不是必需要求。不建议使用这类检测。
产品列表
参考文献
如要继续阅读,请登录或创建帐户。
暂无帐户?